Bre został pierwotnie scharakteryzowany jako sekwencja wiążąca czynnik transkrypcyjny TFIIB. Można wyróżnić dwie specyficzne lokalizacje dla tego motywu. Najczęściej występuje bezpośrednio przed sekwencją TATA. BRE występujący bezpośrednio przed sekwencją TATA jest nazywany BRE upstream i jest tożsamy z pierwotnie zidentyfikowanym motywem BRE. Może on również występować bliżej miejsca startu transkrypcji niż TATA i wtedy jest nazywany BRE downstream. BRE downstream jest odpowiedzialny za tworzenie kompleksu TFIIB-TBP, podobnie jak BRE upstream.
Sekwencja konsensusowa BRE upstream to SSRCGCC. Natomiast BRE downstream posiada sekwencję konsensusową w postaci RTDKKKK. Warty podkreślenia jest fakt, że oba te motywy mogą mieć rożny efekt na wydajność transkrypcji w zależności od typu konkretnego promotora. Motywy BRE mogą zarówno wzmacniać jak i hamować ekspresję danego genu. Trzeba zauważyć, że BRE upstream ma zazwyczaj pozytywny efekt na działanie promotora w kontekście poziomu ekspresji. Natomiast w przypadku BRE downstream te efekty rozkładają się mniej więcej po równo. Zauważono, że negatywne działanie BRE downstream jest skorelowane z obecnością BRE upstream. Niestety dokładne mechanizmy działania tego motywu nie są znane. Możliwe jest antagonistyczne działanie pomiędzy tymi dwoma promotorami, jednak to w dalszym ciągu tylko hipoteza.
Oba występujące motywy BRE są w podobnym stopniu rozpowszechnione w promotorach eukariotycznych. Analiza bioinformatyczna dostępnych promotorów z Eucaryotic Promoter Database wykazała, że około 25% promotorów skupionych zawiera w swojej strukturze motyw BRE. Jednocześnie promotory pozbawione TATA box zawierają również motyw BRE znacznie częściej niż te posiadające TATA box.
Downstream promoter element został zidentyfikowany jako region wiążący TFIID, niezbędny do podstawowej aktywności promotora. Mutacja w jego obrębie może spowodować nawet 50-krotny spadek poziomu transkrypcji. Występuje w wielu promotorach, u Drosophila częstość jego występowania jest porównywalna do występowania TATA box. Jednocześnie jedynie około 16% genów posiada oba te motywy w jednym promotorze.
DPE jest bardzo konserwatywny. Jego miejsce położenia jest stałe, nie różni się u Drosophila i u człowieka - zlokalizowany jest w miejscu od +28 do +33 w odniesieniu do miejsca startu transkrypcji w Inr. Sekwencja konsensusowa tego motywu opisana dla Drosophila to RGWYV. Sekwencja konsensusowa dla człowieka wciąż nie została scharakteryzowana.
Downstream promoter element jest kluczowy dla wiązania TFIID. Czynnik transkrypcyjny TFIID wiąże się jednocześnie do DPE i do Inr. Mutacja w obszarze choćby jednego z nich skutkuje brakiem wiązania się TFIID. Bardzo ważny jest również region pomiędzy tymi motywami. Również tutaj mutacja nawet pojedynczego nukleotydu może spowodować kilkukrotne zmniejszenie poziomu ekspresji danego genu. Można powiedzieć, że w praktyce DPE stanowi jeden ważny regulator ekspresji wspólnie z Inr i regionem pomiędzy nimi. Wciąż nie zbadano dokładnie, jaki jest molekularny mechanizm działania DPE. Z pewnością odpowiada za wiązania dla czynnika transkrypcyjnego TFIID, co jednak ważne - nie wiąże TBP. Prawdopodobnie to podjednostki TAF są odpowiedzialnie za wiązanie się z DPE. Wytypowana dwie najbardziej prawdopodobne podjednostki: TAFII60 i TAFII40. Ogólny mechanizm aktywacji transkrypcji i podstawowe czynniki transkrypcyjne są takie same dla wszystkich skupionych promotorów. Jednak dokładny mechanizm może być inny dla każdego motywu, dlatego skupiono się na poszukiwaniu różnic pomiędzy aktywacją w przypadku BRE i TATA. Biochemiczna analiza doprowadziła do identyfikacji białka NC2/Dr1-Drap1. Jako czynnik transkrypcyjny wzmacnia ono poziom transkrypcji w BRE-zależnych promotorach. Jednocześnie wykazano hamujący wpływ na wydajność transkrypcji w TATA-zależnych promotorach. Co więcej, funkcjonowanie NC2/Dr1-Drap1 prawdopodobnie ma odrębny mechanizm w obu wspomnianych przypadkach, co wskazuje na możliwość wykorzystywania różnych czynników transkrypcyjnych przez odpowiednie motywy.
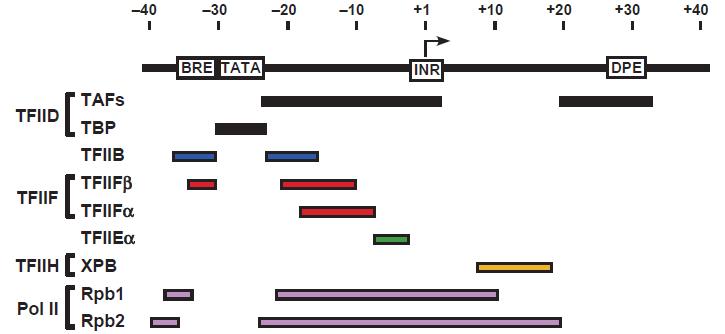
Ilustracja 3: Występowanie czynników transkrypcyjnych w zależności od typu promotora.
Motif ten element (MTE)
Motif ten element obejmuje kilka par zasad w regionie od +18 do +27 w odniesieniu do miejsca startu transkrypcji w Inr u Drosophila. Sekwencja konsensusowa MTE to CSARCSSAAC. Mutacja w obrębie tego regionu może zakłócać funkcjonalność MTE i w związku z tym całego promotora. MTE posiada analogiczne właściwości do DPE w kontekście współpracy z Inr. Tak jak w przypadku DPE, MTE reguluje poziom transkrypcji wspólnie z Inr, a jakiekolwiek zmiany sekwencji pomiędzy tymi motywami skutkują kilkukrotnym zmniejszeniem poziomu ekspresji danego genu. Motif ten element został odnaleziony u Drosophila dzięki połączeniu metod obliczeniowych i eksperymentalnych. MTE nie jest w stanie samodzielnie inicjować wydajnej transkrypcji. Wykazuje natomiast działanie synergistyczne, przede wszystkim wspólnie z TATA i DPE. W związku z tym jego dodatek może kompensować straty wydajności danego promotora w wyniku mutacji w obrębie TATA box lub DPE. To zjawisko stało się inspiracją do zaprojektowania bardzo wydajnego promotora in vitro nazywanego Super Core Promoter.
Downstream core element (DCE)
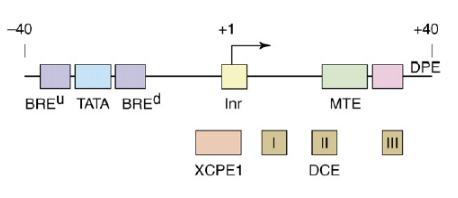
Jest to kolejny ze znanych elementów promotorów skupionych, położony za miejscem startu transkrypcji. Downstream core element występuje często z TATA box. Z kolei jego obecność w promotorach zawierających DPE jest znacznie mniej powszechna. DCE Zbudowany jest z trzech elementów. Pierwszy z nich to sekwencja CTTC w położeniu +6 do +11 w odniesieniu do miejsca startu transkrypcji. Kolejne elementy to sekwencje CTGT i AGC obejmujące obszary +16 to +21 oraz +30 do +34 kolejno. Wszystkie one uczestniczą najprawdopodobniej w wiązaniu podjednostki TAF1. DCE został pierwotnie znaleziony w promotorze genu ludzkiej beta-globiny. W porównaniu z DPE, DCE jest funkcjonalnie unikatowy. Jego działanie można zaobserwować zarówno w połączeniu z TATA box oraz Inr, jak i w promotorach pozbawionych motywu TATA. Stąd wydaje się, że DCE bardziej elastyczny w porównaniu do DPE.
X core promoter element 1 (XCPE1)
Jest to stosunkowo mało popularny i słabo poznany, zwłaszcza w kontekście funkcjonalności, element promotorów skupionych. Na taki stan wiedzy wpływa częstość jego występowania: sekwencję konsensusową XCPE1 w postaci DSGYGGRASM można odnaleźć jedynie w 1% promotorów eukariotycznych. Zazwyczaj jest to skorelowane z brakiem w takim promotorze TATA box. X core promoter element 1 jest położony w obszarze od -8 do +2 w odniesieniu do miejsca startu transkrypcji. Na przekór słabemu rozprzestrzenieniu w promotorach wykazuje bardzo ciekawe właściwości. Jest w stanie samodzielnie aktywować transkrypcję, jednak potrzebuje do tego odpowiednich aktywatorów. Jego funkcjonalność opiera się na współdziałaniu z takimi czynnikami transkrypcyjnymi jak NRF1, NF-1, czy też Sp1. Bez ich udziału jego aktywność jest niemal niezauważalna. W związku w tym X core promoter element 1 jest rozważany jako członek rodziny motywów inicjujących samodzielnie transkrypcję w obrębie wysp CpG.
Wyspy CpG
Promotory rozproszone są znacznie trudniejsze do sklasyfikowania od promotorów skupionych. Ich najlepiej poznanym przedstawicielem są wyspy CpG. Są to genomowe odcinki, w których występuje nadekspresja dinukleotydów CG. To właśnie tutaj najczęściej można odnaleźć promotory rozproszone. Owa struktura ma zastosowanie w wielu typach różnorodnych promotorów. Promotory powiązane z wyspami CpG najczęściej występują w tzw housekeeping gene, czyli genach niezbędnych do prawidłowego metabolizmu i utrzymania podstawowych funkcji życiowych komórki. Zazwyczaj cały funkcjonalny fragment obejmuje obszar od 0,5 do 2 tysięcy par zasad. Na ogół w regionie takiego promotora próżno szukać motywów charakterystycznych dla promotorów skupionych takich jak TATA czy też BRE. Patrząc z perspektywy promotorów skupionych, można powiedzieć, że wyspy CpG zawierają w swojej strukturze kilka słabych promotorów skupionych. To właśnie dlatego charakterystyczną cechą promotorów opartych na tej strukturze jest występowanie wielu miejsc startu transkrypcji.
Ciekawych wniosków dostarczyła analiza czynników transkrypcyjnych w obrębie promotora CpG. Ujawniono wiele miejsc wiązania dla czynnika transkrypcyjnego Sp1, który przyłącza się do charakterystycznych w obrębie wysp CpG tzw. GC box. Ma to bardzo duże znaczenie w kontekście startu transkrypcji. Związanie Sp1 jest w stanie inicjować transkrypcję DNA w połączeniu z Inr przy jednoczesnej absencji TATA box. Dlatego uważa się, że w obrębie CpG może występować wiele motywów Inr, które we współdziałaniu z Sp1 inicjują transkrypcję z równych miejsc w promotorze. Analiza ludzkich genów ujawniła, że około połowa potencjalnych promotorów znajduje się w obszarze wysp CpG. Wciąż jednak mała frakcja wysp CpG może zawierać w swoim obszarze TATA box.
Super Core Promoter
Identyfikacja kolejnych elementów odpowiedzialnych za regulację transkrypcji zrodziła przed naukowcami liczne pytania: A jeśli połączymy je wszystkie razem za sobą za pomocą inżynierii genetycznej? Jak wtedy będzie wyglądał molekularny mechanizm transkrypcji? Jak otrzymać możliwie najsilniejszy promotor? Na te pytania odpowiedzieli Juven-Gershon, Cheng i Kadonaga. W publikacji „Rational design of a super core promoter that enhances gene expression” wykorzystali zjawisko synergii pomiędzy MTE, a innymi elementami promotorów. W ten sposób powstał promotor złożony z motywów TATA box, Inr, MTE oraz DPE, nazwany Super Core Promoter (SCP). W swojej pracy naukowcy opisali sposób projektowania takiego promotora i analizę jego potencjału. SCP złożony ze wspomnianych elementów jest pojedynczym promotorem i jest wyraźnie silniejszy niż promotory cytomegalovirus (CMV) IE1 i adenovirus major late (AdML) zarówno in vitro jak i in vivo. Super Core Promoter jest silnie wiązany przez TFIID i wykazuje tendencję do szybkiego formowania kompleksu transkrypcyjnego. Zaprojektowanie takiego promotora niesie za sobą szereg korzyści. Dzięki temu możliwe będzie biofizyczna analiza wiązania TFIID, biochemiczna analiza całego procesu transkrypcji oraz mechanizmów jego wzmocnienia w komórkach eukariotycznych.
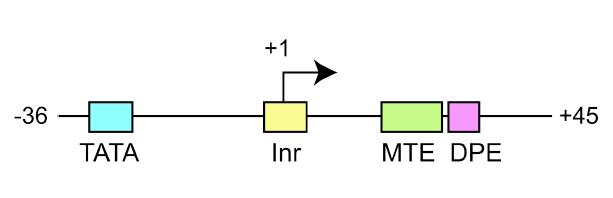
Ilustracja 4: Super Core Promoter
Promotory w obrębie 3'UTR
Większość dobrze poznanych i opisanych promotorów lokalizuje się przed miejsce startu transkrypcji,w obrębie 5' UTR lub nawet w obszarze ulegającym translacji. Jednakże Carninci i współpracownicy w swoich badaniach odkryli zbiory różnorodnych miejsc startu transkrypcji po terminalnej stronie genu, głównie w obszarze 3' UTR. Opracowanie opisuje kompleksowe przeszukiwania startu transkrypcji i analizę wcześniej niezidentyfikowanych odcinków DNA pochodzących z genomu myszy. Zlokalizowali oni granice 181,047 transkryptów z uwzględnieniem wszelkich alternatywnych form wynikających z zastosowania różnych promotorów, alternatywnego slicingu oraz poliadenylacji.
Statystycznie jeden na tysiąc transkryptów jest powiązany z tego typu promotorami. Są one zdolne do inicjowania i regulacji transkrypcji chociaż dokładny mechanizm ich działania i niezbędne czynniki transkrypcyjne wciąż pozostają nieznane. Najbardziej prawdopodobna hipoteza przyjmuje, że odkryte regiony promotorów z odcinków 3' UTR mogą oddziaływać z sekwencjami na nici komplementarnej, formując potencjalne platformy, które mogą wpływać na regulację transkrypcji. Niezależnie od dokładnego mechanizmu działania uzyskane dane z pewnością tworzą podstawę do kompleksowej analizy porównawczej regulacji transkrypcji w różnicowaniu i rozwoju u ssaków.
Ewolucja promotorów
Ewolucja promotorów eukariotycznych przebiega w różnym tempie w zależności od rodzaju genu i miejsca jego ekspresji. Zachodzi ona na skutek akumulacji substytucji oraz delecji lub insercji nukleotydów w obszarze promotorów. Przeprowadzone badania pokazują, że geny tkankowo specyficzne są znacznie bardziej konserwatywne w obszarze promotora w porównaniu z innymi genami. Znajduje to potwierdzenie na przykładzie porównania zmian ewolucyjnych w obrębie promotorów zawierających TATA box oraz wysp CpG. Analiza genomu ludzkiego i mysiego wskazuje, że promotory rozproszone ewoluują znacznie szybciej. Jest to spowodowane faktem, że posiadają one więcej niż jedno miejsce startu transkrypcji. Dzięki temu są znacznie mniej wrażliwe na zmiany w sekwencji DNA, dlatego mutacje w większości przypadków nieznacznie zmieniają poziom ekspresji takiego promotora.
Zmienność w obrębie promotorów jest również bardzo zróżnicowana ze względu na dokładne miejsce mutacji. Badanie ewolucyjne prowadzone na naczelnych, psach, myszach i szczurach ujawniły, że tempo ewolucji jest stosunkowo niskie w obszarze od -50 do -1 par zasad w odniesieniu do miejsca startu transkrypcji. Natomiast dalsza analiza wskazuje na liniowy wzrost tempa ewolucji od nukleotydu -200, czyli im większa odległość od miejsca startu transkrypcji, tym większe tempo ewolucji dla danego promotora. Ma to bardzo duże znaczenie w przypadku promotorów skupionych, gdzie transkrypcja rozpoczyna się w konkretnym miejscu w danej sekwencji. Promotory skupione są zaprojektowane bardzo precyzyjnie, ponieważ odpowiednia architektura motywów sekwencyjnych regulujących transkrypcję gwarantuje inicjację transkrypcji w odpowiednim miejscu i czasie. Jakakolwiek zmiana w sekwencji tych motywów prowadzi do drastycznych zmian w poziomie ekspresji. Przedstawione tezy znalazły potwierdzenie w wielu badaniach przeprowadzonych na materiale genetycznym pochodzącym od ssaków, owadów, roślin i drożdży.
Ciekawych wniosków dostarczyła obserwacja ewolucji promotorów u drożdży. Badano sekwencję promotora zawierającego TATA box dlatego samego genu u różnych gatunków drożdży. W tym eksperymencie wykazano większe różnice pomiędzy pomiędzy gatunkami niż pomiędzy innymi innymi promotorami.
Przyszłość analizy
Na chwilę obecną bardzo dobrze poznane są mechanizm inicjacji transkrypcji w promotorach zawierających TATA box. Celem prowadzonych na szeroką skalę analiz całego genomu jest równie dokładna identyfikacja pozostałych promotorów i poznanie mechanizmów ich działania. Jak pokazują badania, możliwości jest wiele. Kluczowe dla progresu w badaniach okazało się zdefiniowanie miejsca startu transkrypcji i sekwencji wokół niego jako głównych elementów odpowiedzialnych za regulację transkrypcji. To z kolei umożliwia zdefiniowanie czynników transkrypcyjnych zaangażowanych w inicjację i regulację transkrypcji. Takie podejście umożliwiło segregację promotorów na podstawie podobnych mechanizmów pomimo zróżnicowanego występowania. Jak się spodziewano promotory tkankowa specyficzne są bogate w konkretne motywy, który wiążą odpowiednie czynniki transkrypcyjne specyficzne dla danego typu tkanki. Niestety okazuje się, że promotory wcale nie muszą być położone w pobliżu miejsca startu transkrypcji.
Analiza regulacji transkrypcji nie jest wolna od problemów. Duże wyzwanie stanowi powiązanie lokalizacji miejsc startu transkrypcji z takimi zjawiskami jak: metylacja histonów, pozycja nukleosomów, stan acetylacji czy też dostępność miejsc wiążących czynniki transkrypcyjne. Wciąż nierozwiązana pozostaje kwestia występowania miejsc wiązania dla czynników transkrypcyjnych, które to miejsca są bardzo trudne do powiązania z transkrypcją jakiegokolwiek genu. Kolejną trudnością jest powiązanie regionu 5' genu z konkretnym transkryptem, zwłaszcza jeżeli miejsce startu transkrypcji nie jest jeszcze dokładnie znane. Zdarza się, że z jednego miejsca może zachodzić transkrypcja dwóch lub więcej genów.
Aby zrozumieć interakcje pomiędzy elementami regulatorowymi w promotorach i wiążącymi się z nimi białkami regulatorowymi oraz to, jak współdziałają one w regulacji transkrypcji, potrzebne są badania na bardzo szeroką skalę. Nie powinno się ich z pewnością ograniczać tylko do jednego gatunku. Niezbędna jest tutaj współpraca pomiędzy bioinformatykami i biologami molekularnymi. Mnogość odnalezionych promotorów wymaga zastosowania przynajmniej kilku metod analizy w kontekście różnych organów, tkanek i typów komórek. Analiza całego genomu dostarcza doskonałej mapy do poruszania się pomiędzy regionami promotorów i potencjalnych miejsc startu transkrypcji. Bardziej dogłębne badania prowadzą do dokładniejszych opisów kompleksów inicjacyjnych, co tworzy bardziej prawdopodobne hipotezy na temat funkcjonowania elementów regulatorowych. Tak zaprojektowane kompleksy regulatorowe będą mogły wówczas zostać przetestowane eksperymentalnie.
Bibliografia:
1. Sandelin A., et al.: Mammalian RNA polymerase II core promoters: insights from genome-wide studies. Nat Reviews, 2007; 8(6):424-436.
2. Juven-Gershon T., Hsu J.-Y., Theisen J.W., Kadonaga J.T.: The RNA polymerase II core promoter - the gateway to transcription. Current Opinion in Cell Bio, 2008; 20 (3):53-259.
3. Butler J.E.F., Kadonaga J.T.: The RNA polymerase II core promoter: A key component in the regulation of gene expression. Genes and Development, 2002; 16 (20):2583-2592.
4.Tokusumi Y., et al.: The new core promoter element XCPE1 (X core promoter element 1) directs activator-, mediator-, and TATA-binding protein-dependent but TFIID-independent RNA polymerase II transcription from TATA-less promoters. Mol and Cell Bio, 2007; 27 (5):1844-1858.
5.Tirosh I., Weinberger A., Carmi M. & Barkai N.:A genetic signature of interspecies variations in gene expression. Nature Genet, 2006; 38:830–834.
6. Carninci P., et al. The transcriptional landscape of the mammalian genome. Science, 2005; 309:1559–1563.
7. Juven-Gershon T, Cheng S, Kadonaga JT: Rational design of a super core promoter that enhances gene expression. Nat Methods 2006, 3:917-922.
8.Hahn S.: Structure and mechanism of the RNA polymerase II transcription machinery. Nat Struct Mol Biol, 2004; 11(5): 394-403
9. Deng, W., Roberts, S.G.E.:TFIIB and the regulation of transcription by RNA polymerase II. Chromosoma, 2007; 116(5):417-429
10.Lewis, B.A., Kim, T.-K., Orkin, S.H.: A downstream element in the human β-globin promoter: Evidence of extended sequence-specific transcription factor IID contacts. Proceedings of the National Academy of Sciences of the USA, 2000; 97 (13):7172-7177.
11.Burke T.W., Kadonaga J.T.: The downstream core promoter element, DPE, is conserved from Drosophila to humans and is recognized by TAF(II)60 of Drosophila. GENES & DEVELOPMENT 1997; 11(22):3020-3031.
opracował: Wojciech Antosz









Recenzje