
Projekt dot. osób z niepełnosprawnościami na studiach podyplomowych
Uczelnie zyskają większą elastyczność w wykorzystaniu środków.
|
Zamknij X
|
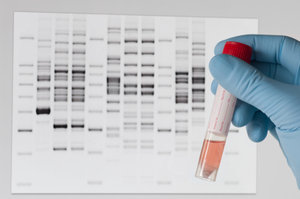 Niepełnosprawność intelektualna dotyczy 2-3 proc. populacji, zaburzenia autystyczne – 1 proc., padaczka – również 1 proc. Wrodzone wady serca stwierdza się u 5-8 na 1 tys. żywych urodzeń, stanowią one 1/3 wszystkich wad wrodzonych i są najczęstszą przyczyną śmierci niemowląt. Zapotrzebowanie na diagnostykę tych patologii jest duże. Umożliwia ją m.in. oryginalne narzędzie polskiego projektu – specjalna macierz.
Niepełnosprawność intelektualna dotyczy 2-3 proc. populacji, zaburzenia autystyczne – 1 proc., padaczka – również 1 proc. Wrodzone wady serca stwierdza się u 5-8 na 1 tys. żywych urodzeń, stanowią one 1/3 wszystkich wad wrodzonych i są najczęstszą przyczyną śmierci niemowląt. Zapotrzebowanie na diagnostykę tych patologii jest duże. Umożliwia ją m.in. oryginalne narzędzie polskiego projektu – specjalna macierz.
Uczelnie zyskają większą elastyczność w wykorzystaniu środków.

Mogą nieznacznie opóźnić pojawienie się ich wiosną.

Wynika z badania, które publikuje „Blood Red Cells & Iron".

Informuje czasopismo „Alcohol”.

Nawet, gdy matka mówi w innym języku niż jej ojczysty.

Informuje czasopismo „Trends in Neurosciences”.

Wykazały analizy Zespołu Badań nad Językiem i Humorem.

Unijne fundusze na naukę wciąż za mało wykorzystywane przez Polaków
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje