Aktualnie znanych jest wiele różnych metod badania kwasów nukleinowych. Zazwyczaj opierają się one albo na przeprowadzenie charakterystycznych barwnych reakcji chemicznych, bądź na pomiarze spektrofotometrycznym. Bardzo często do rozdziału, a także do identyfikacji i oczyszczania kwasów nukleinowych stosuje się elektroforezę w żelu agarozowym. Słowa kluczowe: pomiar spektrofotometryczny, orcyna, furfural, reakcja barwna, metoda orcynolowa, metoda Dischego, elektroforeza w żelu agarozowym, pomiar nadfiolecie.
Wolne pentozy, fosfopentozy bądź związane w nukleotydach purynowych w kwasach nukleinowych, w wyniku ogrzewania ze stężonym kwasem solnym (HCl) mają zdolność przechodzenia w furfural. Powstały w wyniku reakcji furfural tworzy z orcyną (odczynnik analityczny stosowany przy oznaczaniu cukrów) kompleks o trwałej zielonej barwie. Reakcja katalizowana jest przez jony Fe3+ . Maksimum absorpcji powstałego barwnego kompleksu przypada przy długości fali równej 610 nm. W takich warunkach deoksyryboza reaguje ok. 10-krotnie słabiej od rybozy, w związku z czym odczyn ten dla DNA wypada w postaci ujemnej. Reakcji tej nie daje ryboza, która związana jest z zasadami pirymidynowymi lub z amidem kwasu nikotynowego [4].
Oznaczanie RNA metodą orcynolową [1], [2].W metodzie tej w obecności soli żelaza (III) (soli żelazowych), furfural, który powstaje w rybozy w środowisku HCl daje z oryną kompleks o barwie zielonej [1], [2].
W celu przeprowadzenia doświadczenia, do probówki wirówkowej skalowanej na 10 ml należy dodać 2 ml roztworu rybonukleinianu magnezowego (tj.: lekko zasadowy roztwór wodny o stężeniu ok. 1 mg/ml) oraz 10-krotnie mniejszą objętość 1 M roztworu KOH. Probówkę z odczynnikami umieścić we wrzącej łaźni wodnej na 15 minut, od czasu do czasu mieszając jej zawartość. Po inkubacji, próbkę oziębić i zobojętnić za pomocą 60% roztworu HClO4, po czym dodać 60% roztwór HClO4 do stężenia 3%. Probówkę należy umieścić w lodzie , inkubować w lodzie przez 15 minut, a następnie odwirować osad zawierający KClO4 i ewentualny DNA. Otrzymany po wirowaniu supernatant przenieść do kolby miarowej o pojemności 25 ml. Osad przemyć 2 razy za pomocą 1 ml H2O w celu pełnego wyekstrahowania rybo nukleotydów z osadu i dołączyć do cieczy (supernatant) w kolbie. Wtedy, zawartość kolbki uzupełnić do kreski wodą, po czym starannie wymieszać [1], [2].
Do nowej, suchej probówki pobrać ilościowo 2 ml hydrolizatu, dodać do niego 1 ml wody oraz 3 ml odczynnika orcynolowego (tj.: 1 g oczyszczonego orcynolu rozpuścić w 100 ml stężonego roztworu HCl zawierającego 0,5 g FeCl3. W celu oczyszczenia handlowego orcynolu należy rozpuścić go w gorącym benzenie, wytrząsać z węglem aktywnym i po usunięciu węgla wykrystalizować orcynol po dodaniu heksanu). Zawartość próbki dokładnie wymieszać, po czym umieścić we wrzącej łaźni wodnej na 20 minut. Równolegle, w identyczny sposób należy przygotować próbę kontrolną na odczynniki oraz próbę zawierającą wzorcowy roztwór RNA [1], [2].
W probówkach zawierających RNA powstaje trwałe, zielone zabarwienie kompleksu furfuralu z orcyną. Po ostudzeniu przygotowanych probówek należy odczytać wartości absorbancji przy długości fali równej λ= 660 nm (jest to maksimum pochłaniania przez utworzony kompleks) w fotokolorymetrze- w odniesieniu do próby kontrolnej [1], [2].
Zawartość RNA znajdującą się w próbce badanej obliczyć przez porównanie absorbancji próby badanej z absorbancją próby wzorcowej. Jako wzorca w ilościowych oznaczeniach RNA używa się roztwory RNA o znanej zawartości fosforu RNA (tj. RNA-P), natomiast nigdy nie stosuje się jako wzorca wolnej rybozy. Wynik należy podać w mg i μg zawartość RNA w 1 ml oznaczonego roztworu rybonukleinianu magnezowego, a także podać procentową zawartość RNA w otrzymanym preparacie nukleinianu magnezowego [1], [2].
Rys. http://www.food-info.net/pl/colour/maillard.htm [3].W reakcjach przeprowadzanych z orcyną , barwne kompleksy daje tylko ryboza, która uwolniona jest z nukleotydów i nukleozydów purynowych. Ryboza, która połączona jest z zasadami pirymidynowymi nie wchodzi w reakcję (nie daje barwnego kompleksu) [1], [2].
Z orcyną (poza rybozą) może reagować także 2-deoksyryboza, która daje 10-krotnie słabsze zabarwienie niż ryboza. Podczas oznaczania ilościowego RNA w obecności Dna, zawsze trzeba mieć na uwadze wprowadzenie poprawek na zawartość DNA [1], [2].
Próba Biala z orcyną na pentozy [2].W wyniku działania na pentozy kwasem solnym powstaje furfural. Ten następnie w połączeniu z floroglucyną tworzy kompleks o zabarwieniu wiśniowym. Analogicznie dzieje się w przypadku wykorzystania np. kwasów uronowych, które w zaistniałych warunkach ulęgają procesowi dekarboksylacji [2].
By przeprowadzić wykrywanie pentoz metodą Biala, do 1 ml roztworu pentozy (lub heksozy) należy dodać 2 ml stężonego roztworu kwasu solnego (HCl) oraz 4 krople 2% etanolowego roztworu floroglucyny. Otrzymaną mieszaninę należy ogrzać, w wyniku czego roztwór zmienia barwę na wiśniowofioletowy kolor. W przypadku wykrywania heksoz powstaje żółte lub brązowe zabarwienie roztworu [2].
Rozdział kwasów nukleinowych za pomocą elektroforezy w żelu agarozowym [7].Do jednej z głównych metod identyfikacji, rozdzielania i oczyszczania kwasów nukleinowych zalicza się elektroforezę w żelu agarozowym. Ze względu na fakt, że cząsteczka DNA w środowisku obojętnym i alkaicznym posiada ładunek ujemny, tak więc umieszczona w polu elektrycznym wędruje w kierunku anody. Ponadto, DNA posiadające tą samą masę cząsteczkową a różną konformację, mają różną ruchliwość elektroforetyczną. Istnieje prosta zależność, że im dłuższa jest cząsteczka DNA lub RNA , tym dłużej zachodzi jej rozdział w porach żelu. By otrzymać optymalny rozdział DNA o wielkości większej niż 2 kpz należy prowadzić elektroforezę w polu elektrycznym o natężeniu nie większym niż 5V/cm. Powyżej tego napięcia rozdział cząsteczki zachodzi odwrotnie proporcjonalnie do logarytmu ciężaru cząsteczkowego. Zachowanie cząsteczek DNA lub RNA w podczas rozdziału elektroforetycznego częściowo zależy od temperatury i nie zależy od składu zasad azotowych [7].
By po rozdziale elektroforetycznym możliwe było uwidocznienie DNA w żelu, stosuje się bromek etydyny (EtBr), który ma zdolność interkalowania pomiędzy sąsiadujące pary dwuniciowego DNA. Powinowactwo bromku etydyny do jednoniciowego DNA jest znacznie mniejsze. Podczas dodania bromku etydyny, należy mieć na uwadze, że jego obecność żelu agarozowym zmniejsza ruchliwość elektroforetyczną o około 15%. Często stosowanym barwnikiem do uwidaczniania kwasów nukleinowych w żelu jest SYBR Green, który charakteryzuje się mniejszą czułością barwienia dwuniciowego DNA niż EtBr (ok. 25 razy) [7].
Odróżnianie DNA od RNA- wykrywanie DNA metodą Dischego [4].W skład DNA wchodzą nukleotydy zawierające 2-deoksy-D-rybozę, z kolei RNA budują nukleotydy zawierające D-rybozę. Dzięki istnieniu różnych pentoz w tych dwóch kwasach nukleinowych, możliwe jest odróżnienie preparatu DNA od RNA za pomocą różnych metod chemicznych [4].
Metoda wykrywania DNA opiera się na tym, że w środowisku kwaśnym (stężone H2SO4 lub CH3COOH) deoksyryboza występującą w stanie wolnym lub związanym w nukleotydach purynowych, tworzy z difenyloaminą produkt kondensacji o charakterystycznym niebieskim zabarwieniu. Pojawienie się barwnej reakcji, jest konsekwencją powstania aldehydu hydroksylewulinowego z deoksyrybozy w wyniku działania kwasu siarkowego i octowego. Następnie, aldehyd ten ulega kondensacji z difenyloaminą , w wyniku czego powstaje produkt o barwie niebieskiej. Maksimum absorpcji powstałego produktu przypada przy λ=600 nm. Ponadto, oprócz deoksyrybozy, reakcji tej podlega także kwas N-acetyloneuraminowy- produkt powstały w środowisku obojętnym bądź zasadowym ma żółtą barwę [4].
W celu przeprowadzenia doświadczenia należy przygotować w statywie 4 probówki, a następpnie do każdej z nich odmierzyć po 0,5 ml odpowiednich roztworów tj.: kwasu nukleinowego nr 1 do pierwszej probówki, kwasu nukleinowego nr 2 do drugiej probówki, deoksyrybozy do trzeciej probówki oraz wody destylowanej do czwartej probówki (będzie o próba ślepa). Następnie, do wszystkich probówek wprowadzić po 1 ml odczynnika Dischego (tj.: 1% difenyloamina w H2SO4 i lodowaty CH3COOH). Całość dobrze wymieszać. Probówki z analizowanymi próbkami wstawić do wstawić do wrzącej łaźni wodnej- inkubować przez 10 minut. Po inkubacji przeprowadzić identyfikację próbek na podstawie charakterystycznego zabarwienia [4].
Oznaczanie zawartości DNA metodą difenyloaminową (próba na deoksyrybozę) [1], [2].Deoksyryboza, która jest składnikiem kwasu deoksyrybonukleinowego (DNA), z odczynnikiem difenyloaminowym zawierającym stężony kwas CH3COOH, daje niebieskie zabarwienie. Dzięki temu możliwe jest jej odróżnienie od rybozy [1], [2].
Jako materiał do przeprowadzenia doświadczenia może być użyte 3 g grasicy, zhomogenizowanej z 5-krotną objętością zimnego 10% roztworu CCl3COOH w homogenizatorze nożowym. Powstały homogenat należy przenieść ilościowo do probówki wirówkowej (o objętości 50 ml), umieszczonej w łaźni lodowej, mieszać przez 10 minut. Po tym czasie homogenat odwirować, powstały po wirowaniu supernatant usunąć, a osad przemyć 10-krotną objętością zimnego 10% roztworu CCl3COOH i ponownie odwirować. Zastosowanie zimnego roztworu CCl3COOH powoduje, że ekstrahowane są z tkanki małocząsteczkowe związki frakcji rozpuszczalnej (tj. nukleotydy, aminokwasy, estry fosforanowe, fosforany nieorganiczne), z kolei w osadzie pozostają białka, wielocukry i kwasy nukleinowe [1], [2].
Do osadu należy dodać 10 ml 0,6 M roztworu HClO4, po czym ogrzewać mieszaninę w łaźni wodnej w temperaturze 90˚C przez 15 minut stale mieszając. Dzięki ogrzewaniu w wysokiej temperaturze w roztworze HClO4 dochodzi do uwolnienia kwasów nukleinowych (RNA i DNA) od białka, ulegają hydrolizie do związków rozpuszczalnych w kwasach i przechodzą do roztworu. Po ostudzeniu, próbki odwirować (osad zawierający białko), zaś supernatant przenieść ilościowo do kolbki miarowej o pojemności 25 ml. Otrzymany osad przemyć 2-krotnie 5 objętościami 0,6 M roztworu HClO4 i supernatant dołączyć do roztworu znajdującego się w kolbce, następnie zawartość kolbki uzupełnić do kreski wodą, a całość dokładnie wymieszać [1], [2].
Otrzymany ekstrakt kwasów nukleinowych (1 ml) przenieść do probówki. Do niego dodać 1 ml wody i 2-krotną objętość (4 ml) odczynnika difenyloaminowego (tj.: 1 g oczyszczonej difenyloaminy rozpuścić w 100 ml lodowatego CH3COOH z dodatkiem 2,75 ml stężonego roztworu H2SO4. W celu oczyszczenia difenyloaminy należy handlowy preparat przekrystalizować z gorącego heksanu). Zawartość probówki dokładnie wymieszać, po czym umieścić we wrzącej łaźni wodnej na 10 minut. Równolegle przygotować próbę kontrolną na odczynnika a także próbę z wzorcowym DNA o znanej zawartości fosforu DNA tj. DNA-P (wzorcowy DNA o stężeniu 20 μg DNA-P w 1 ml) [1], [2].
W probówkach, w których jest DNA powstanie trwałe, niebieskie zabarwienie. Absorbancję oznaczanej próby wzorcowej należy odczytać w odniesieniu do próby kontrolnej. Pomiaru dokonać przy długości fali równej λ= 600 nm (j. maksimum pochłaniania przez utworzony kompleks barwny) [1], [2].
Zawartość DNA w próbce badanej obliczyć przez porównanie absorbancji próby badanej z absorbancją próby wzorcowej- zawartość DNA podać w mg/g świeżej tkanki [1], [2].
 Zdjęcie: deoksyryboza, http://wdict.net/pl/gallery/deoksyryboza/ [6].Reakcje barwne składnika cukrowego [5].
Zdjęcie: deoksyryboza, http://wdict.net/pl/gallery/deoksyryboza/ [6].Reakcje barwne składnika cukrowego [5].Metody te opierają się na mniej lub bardziej specyficznych odczynach barwnych, które są charakterystyczne dl pentozy lub deoksypentozy. Metody te zostały dość szczegółowo omówione przez Dische’go. W reakcjach tych pentozy nie utrudniają oznaczania deoksypentozy i na odwrót. Jednakże większość tych metod nie może być stosowana w obecności białek, a to dlatego, że powstaje wtedy furfural, który znacznie utrudnia oznaczenie. W związku z tym, przed zastosowaniem metod kolorymetrycznych ważne jest, by możliwie jak najdokładniej uwolnić kwasy nukleinowe od pozostałości białek tkankowych [5].
Bardzo prostą metodę przeprowadzeni opisanej powyżej reakcji przedstawili Davidson i Waymouth. Przeprowadzając reakcję w ciągu kilku godzin w 30˚C oraz w obecności aldehydu octowego, możliwe jest znaczne zwiększenie czułości reakcji – w porównaniu z pierwotną metodą przeprowadzaną w 100˚C przez kilka minut. W metodzie tej obniża się także wrażliwość próby na obecność substancji przeszkadzających [5].
Ceriotti opisał także bardzo dogodną a dodatkowo bardziej czułą reakcję barwną przeprowadzaną z wykorzystaniem indolu. Przedstawiona przez niego metoda nadaje się do oznaczeń ilościowych kwasów nukleinowych [5].
Podobna, mniej skomplikowana lecz także mniej znana metoda oznaczania deoksypentozy, opiera się na ogrzewaniu DNA z cysteiną i kwasem siarkowym, w wyniku czego powstaje różowe zabarwienie [5].
Oznaczanie puryn i pirymidyn przez pomiar absorpcji w nadfiolecie [5].Po oddzieleniu DNA od RNA za pomocą metody Schmidta i Thannhausera bądź Ogura i Rosena, możliwe jest przeprowadzenie łatwego oznaczenia ilości puryn i pirymidyn , a dzięki temu można oznaczyć ilość kwasów nukleinowych w każdej frakcji. W tym celu przeprowadza się pomiar ekstynkcji roztworu w świetle nadfioletowym przy użyciu spektrofotometru kwarcowego. Podczas sporządzania wyciągów do tego typu pomiarów zaleca się stosowanie kwasu kwasu nadchlorowego zamiast trójchlorooctowego, a to ze względu na fakt, że kwas trójchlorooctowy wykazuje silną absorpcję w nadfiolecie- szczególnie przy 260 nm [5].
W swoich doświadczeniach, Logan, Mannell i Rossiter analizując tkankę nerwową wykorzystali zalety metody Schmidta i Thannhausera oraz Schneidera. W związku z tym, frakcję P-DNA oddzielili pierwszą z metod, następnie ekstrahowali DNA gorącym kwasem trójchlorooctowym, po czym oznaczali ekstynkcję przy 268,5 nm . Do ogólnego wyodrębnienia frakcji kwasów nukleinowych użyli metody Schneidera, po czym oznaczali ekstynkcje także przy 268,5 nm. Ilość RNA oznaczali na podstawie różnicy w przeprowadzonych oznaczeniach [5].
Zasada pomiaru gęstości optycznej w nadfiolecie (zamiast reakcji barwnych lub oznaczania fosforu) była wprowadzana także przez innych badaczy. Metoda ta jest godna polecenia ze względu na fakt, że jest szybka i prosta w wykonaniu, a także swoista. Jednakże nie jest metodą wolną od wad, ponieważ stosowanie jej wymaga przyjęcia umownej wartości dla ekstynkcji ε (P), co z kolei nie jest zupełnie ścisłe dla kwasów nukleinowych występujących w niektórych tkankach. Kwasy nukleinowe w wyniku ogrzewania z kwasami wykazują zmiany w krzywych absorpcji. Dzięki badaniom Logan’a i wsp. udało się stwierdzić, że krzywe absorpcyjne dla równych stężeń DNA i RNA przecinają się przy λ=268,5 nm. Zauważono również, że właśnie przy tej długości fali obydwa rodzaje kwasów nukleinowych (DNA i RNA) mają wartość ε (P)= 9850 [5].
Należy zaznaczyć, że sam kwas trójchlorooctowy ma dużą zdolność absorpcji w świetle nadfioletowym, którą uwzględnia się w obliczeniach dzięki zastosowaniu odpowiednich ślepych prób. Właśnie na tych założeniach opiera się jedna z modyfikacji metody Schmidta i Thannhausera, która została wprowadzona przez Scott’a, Fraccastoro i Taft’a, do oznaczania mikrogramowych ilości kwasów nukleinowych w skrawkach tkanek [5].
Autor: Lidia KoperwasLiteratura:[1]. Schneider W.C., 1957. Determination of nucleic acids in tissues by pentose analysis. [in:]Methods in enzymology, vol. 3. S.P. Colowick, N.O.Kaplan, Academic Press, New York, .s. 680-684
[2]. Kłyszejko-Stefanowicz L, 2003. Ćwiczenia z biochemii. Wydawnictwo Naukowe PWN, 2003, s. 272, 355-359
[3]. http://www.food-info.net/pl/colour/maillard.htm
[4]. [http://biochigen.slam.katowice.pl/praktikum/013.pdf]
[5]. Davidson J.N, F.R.S, 1969. The Biochemistry of the Nucleic Acids, Six Edition , 1969; s. 112-115.
[6]. http://wdict.net/pl/gallery/deoksyryboza/
[7].http://www.pg.gda.pl/chem/Katedry/Leki/Dydaktyka/Kultury/lab/Porownanie_ilosci_i_%20jakosci_DNA_wyizolowanego_z_komorki_roslinnej_i_zwierzecej.pdf


 Wolne pentozy, fosfopentozy bądź związane w nukleotydach purynowych w kwasach nukleinowych, w wyniku ogrzewania ze stężonym kwasem solnym (HCl) mają zdolność przechodzenia w furfural. Powstały w wyniku reakcji furfural tworzy z orcyną (odczynnik analityczny stosowany przy oznaczaniu cukrów) kompleks o trwałej zielonej barwie. Reakcja katalizowana jest przez jony Fe3+ . Maksimum absorpcji powstałego barwnego kompleksu przypada przy długości fali równej 610 nm. W takich warunkach deoksyryboza reaguje ok. 10-krotnie słabiej od rybozy, w związku z czym odczyn ten dla DNA wypada w postaci ujemnej. Reakcji tej nie daje ryboza, która związana jest z zasadami pirymidynowymi lub z amidem kwasu nikotynowego [4].
Wolne pentozy, fosfopentozy bądź związane w nukleotydach purynowych w kwasach nukleinowych, w wyniku ogrzewania ze stężonym kwasem solnym (HCl) mają zdolność przechodzenia w furfural. Powstały w wyniku reakcji furfural tworzy z orcyną (odczynnik analityczny stosowany przy oznaczaniu cukrów) kompleks o trwałej zielonej barwie. Reakcja katalizowana jest przez jony Fe3+ . Maksimum absorpcji powstałego barwnego kompleksu przypada przy długości fali równej 610 nm. W takich warunkach deoksyryboza reaguje ok. 10-krotnie słabiej od rybozy, w związku z czym odczyn ten dla DNA wypada w postaci ujemnej. Reakcji tej nie daje ryboza, która związana jest z zasadami pirymidynowymi lub z amidem kwasu nikotynowego [4].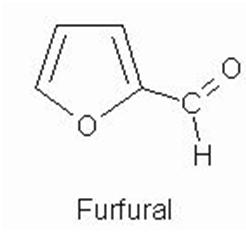

Recenzje