

|
Zamknij X
|
 Enzymy restrykcyjne, określane także jako restryktazy czy endonukleazy restrykcyjne, są enzymami izolowanymi z bakterii lub sinic (rzadziej z promieniowców), które zdolne są do rozpoznawania specyficznych sekwencji w DNA, a także do przecinania dwuniciowej cząsteczki DNA [8].
Enzymy restrykcyjne, określane także jako restryktazy czy endonukleazy restrykcyjne, są enzymami izolowanymi z bakterii lub sinic (rzadziej z promieniowców), które zdolne są do rozpoznawania specyficznych sekwencji w DNA, a także do przecinania dwuniciowej cząsteczki DNA [8].Enzymy klasy II:
Enzymy klasy III:
W wyniku trawienia określonej cząsteczki DNA danym enzymem restrykcyjnym powstaje zawsze taka sama liczba fragmentów określonej wielkości. Liczbę i wielkość uzyskiwanych fragmentów DNA ustala się poddając strawiony DNA elektroforezie w żelach agarozowych lub akryloamidowych. Analizując produkty trawienia różnymi enzymami restrykcyjnymi (stosowanymi pojedynczo, w kombinacjach, oraz w ilościach wystarczających lub niewystarczających do pełnego strawienia preparatu) możliwe jest ustalenie wzajemnego położenia i odległości pomiędzy sekwencjami rozpoznawanymi przez te enzymy. Tak więc możliwe jest sporządzenie mapy restrykcyjnej danej cząsteczki DNA [8], [4].
Wszystkie enzymy po trawieniu dwuniciowego DNA zostawiają grupę fosforanową na końcu 5’, zaś grupę hydroksylową na końcu 3’. Aktywność enzymów restrykcyjnych zależy od stężenia soli (chlorku sodu – NaCl), która zawarta jest w buforze. W zależności od rodzaju, niektóre enzymy wykazują maksimum swojego działania przy wysokim stężeniu soli (150mM), zaś inne przy niskim [4].
Dane dotyczące wymaganego stężenia soli zamieszczają katalogi firm produkujących enzymy (np. New England Biolabs, Fermentas). Zawsze wskazane jest stosowanie buforów zalecanych przez producenta danego enzymu. W przypadku, gdy DNA ma być strawiony dwoma różnymi enzymami restrykcyjnymi, które wymagają różnych buforów, należy najpierw przeprowadzić trawienie w buforze o niższym stężeniu soli, a następnie podwyższyć stężenie soli w mieszaninie reakcyjnej poprzez dodanie odpowiedniej objętości stężonego roztworu NaCl. Jeżeli DNA ma być trawiony kilkoma enzymami restrykcyjnymi, możliwe jest przeprowadzenie jednoczesnej reakcji dla wszystkich enzymów, jednak pod warunkiem, że znajdzie się bufor, w którym wszystkie enzymy będą efektywnie działać [7], [8], [10].
Na ogół różne enzymy restrykcyjne rozpoznają różne sekwencje DNA. Znanych jest jednak wiele przykładów enzymów restrykcyjnych, które izolowane są z odrębnych organizmów, a mimo to rozpoznają takie same sekwencje DNA. Przykładem może być np. enzymy Sau3AI oraz MboI, które rozpoznają sekwencję 5’GATC3’. Enzymy cechujące się takimi właściwościami nazywamy izoschizomerami. Izoschizomery mogą ciąć rozpoznawaną sekwencję w różny sposób [np. MboI (G/TAC) i DpnI (GA/TC) [9], [10].
Bufory do trawienia
Bufory stosowane do trawienia przygotowane są w postaci 10-krotnie stężonych roztworów, zaś do trawienia nie należy dodawać więcej enzymu niż 1/10 objętości mieszaniny reakcyjnej. Stężone preparaty enzymów należy zawsze rozcieńczać w odpowiednim buforze- nigdy w wodzie ze względu na możliwość denaturacji [7].
Z dostępnych na rynku enzymów, niektóre mają dodatkowe aktywności, które pojawiają się w pewnych warunkach trawienia (zjawisko to określane jest jako aktywność „star”). Informacje o takiej aktywności zawsze podawane są przez producentów enzymów restrykcyjnych w dołączonych do opakowania ulotkach. Sporządzając mieszaninę reakcyjną, należy pamiętać, że enzym dodaje się zawsze na końcu, a dodawane enzymy bezwzględnie muszą być trzymane w temperaturze -20°C, wtedy też wykazują swoją stabilność. Najlepiej jak enzymy przechowywane są w niskiej temperaturze w buforze zawierającym 50% glicerol [7].
Warunki trawienia muszą być takie same jak zalecane w katalogach producentach. Trawienie enzymami restrykcyjnymi można zatrzymać przez dodanie roztworu EDTA, a następnie grzanie mieszaniny przez 10 do 15 minut w temperaturze sięgającej 65-70°C lub ekstrakcję fenolem. Co ważne, należy pamiętać, że niektóre z dostępnych enzymów są termo stabilne – tj. nie ulegają termicznej inaktywacji [8].
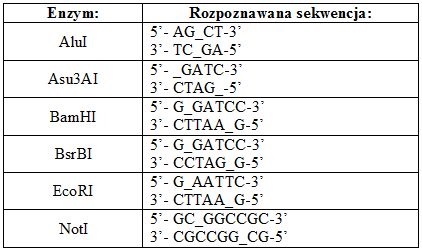
Zastosowanie enzymów restrykcyjnych:
Zdjęcie: http://microbio.gumed.edu.pl/materialy/biol_mol/Enzymy_restrykcyjne.pdf
Trawienie DNA enzymami restrykcyjnymi [10].
Zazwyczaj, inkubację preparatów DNA z enzymami restrykcyjnymi prowadzi się w temperaturze 37oC, w czasie 1 godziny lub dłużej. Przerwanie reakcji (trawienia) można przeprowadzić przez termiczną inaktywację danego enzymu, którą to przeprowadza się w 65oC przez 15 minut. Innym sposobem przerwania trawienia jest dodanie do próbki EDTA o pH=8.0 do końcowego stężenia równego 10 mM (chelatacja jonów magnezu) [6], [10].
Jednostkę enzymu definiuje się jako taką ilość enzymu, która jest w stanie kompletnie strawić 1 µg DNA w odpowiednim buforze i optymalnej temperaturze w objętości 20 uL w czasie 1 godziny.
Wykonanie:
Roztwór zawierający ok. 1,2 do 1 µg DNA należy umieścić w probówce z dodatkiem wody (do objętości równej 18 µl). Następnie, do próbki dodać 2 µl odpowiedniego buforu do trawienia(10x stężonego). Jako bufor trawiący można zastosować 2x stężony bufor KGB (kalium glutamate buffer: 200 mM glutaminian potasu - 50 mM Tris/octan (o pH=7.5) - 20 mM (CH3COO)2Mg - 100 µg/ml albumina z surowicy cieląt - 1 mM 2-merkaptoetanol). Całość delikatnie wymieszać (przez uderzanie probówki palcem).
Po wymieszaniu do próbki dodać 1-2 jednostki enzymu restrykcyjnego, wymieszać, inkubować w optymalnej temperaturze przez czas zalecany przez producenta. Reakcję trawienia zatrzymać przez dodanie EDTA (0,5 M roztwór EDTA o pH=8.0) [7], [10].
Analiza strawionego DNA:
Strawiony enzymami restrykcyjnymi DNA można poddać analizie wieloma sposobami, choć najbardziej znana i najczęściej wykorzystywana jest elektroforeza w żelach agarozowych.
Stosując DNA prokariotyczne wraz z odpowiednio dobranym enzymem, w wyniku trawienia i po rozdziale otrzymuje się kilka pasm odpowiadających fragmentom restrykcyjnym. Preparat kontrolny, tj. taki, który nie jest potraktowany enzymem daje tylko jedno pasmo. W przypadku DNA eukariotycznego w wyniku trawienia enzymem restrykcyjnym otrzymuje się zazwyczaj przesunięcie całego pasma odpowiadającego danemu DNA w stronę odpowiadającą mniejszej ruchliwości elektroforetycznej [7].
Autor: Lidia Koperwas
Literatura:
[1]. Lewiński W., "Genetyka" (Wydawnictwo Operon, 2001);
[2]. Brown T.A., "Genomy" (Wydawnictwo Naukowe PWN, 2001);
[3]. "Genetyka molekularna" praca zbiorowa pod red. Piotra Węgleńskiego (Wydawnictwo Naukowe PWN, 2000).
[4]. http://microbio.gumed.edu.pl/materialy/biol_mol/Enzymy_restrykcyjne.pdf
[5]. http://www.genowo.website.pl/enzymy_restrykcyjne.php
[6]. http://biotechnologia.pl/biotechnologia-portal/info/biotechnologia/32_opracowania/229373,enzymy_restrykcyjne.html
[7]. Kłyszejko-Stefanowicz L, 2003. Ćwiczenia z biochemii. Wydawnictwo Naukowe PWN, 2003, s. 435 – 437
[8]. Genetyka Molekularna, materiały do zajęć laboratoryjnych, 2003. Skrypt przygotowany przez pracowników Zakładu Genetyki UW.
[9]. https://mailattachment.googleusercontent.com/attachment/?ui=2&ik=6bef97507a&view=att&th=13f8768ee45d3012&attid=0.3&disp=inline&realattid=f_higfffww2&safe=1&zw&saduie=AG9B_P8OSjsvMs-ZXGQd5UvJTKLx&sadet=1372666515503&sads=JXJ5sRu8hm0R7oC_e7nmfbpem1k
[10]. Sambrook J i wsp., 1989. Molecular cloning. A laboratory manual . Book 3. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, 5.31 – 5.32
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje