

|
Zamknij X
|
Rybonukleazy (RNazy) są enzymami,
których główną funkcją jest rozkładanie wiązań fosfodiestrowych w kwasach
rybonukleinowych (RNA). Obecność tych
specyficznych enzymów stwierdzono u wszystkich organizmów, przy czym w zależności od gatunku różnią się one specyficznością oraz mechanizmem działania
enzymatycznego [1]. Rybonukleazy występują także w skórze ludzkiej, gdzie
niektóre RNazy z ludzkiego naskórka wykazują tylko aktywność hydrolityczną,
podczas gdy inne uważane są za istotne w procesie przyczepności i złuszczania
keratynocytów [6].
Scharakteryzowano dwie główne klasy rybonukleaz, a mianowicie:
Jako enzymy rybonukleazy pełnią w komórce różnorodne funkcje. I tak przypisuje się im udział w procesie dojrzewania prekursorów kwasów nukleinowych oraz w rozkładzie RNA. Ponadto, Rnaza I uczestniczy w układzie obronnym organizmu. Choć specyficzność Rnaz wiąże się z jednoniciowymi fragmentami kwasu rybonukleinowego, znana jest też tzw. Rnaza H, która atakuje hybrydy RNA-DNA.
Obok typowych Rnaz występują też tzw. rybonukleazy cyklizujące, które rozkładają wiązania fosfodiestrowe z wytworzeniem pośredniego produktu cyklicznego. Reakcja zachodzi w dwóch etapach:
W komórkach roślinnych występują rybonukleazy roślinne, które również rozkładają wszystkie rodzaje RNA- najłatwiej jednak atakują mRNA. Aktywność enzymu zależy od bardzo wielu warunków i tak:
Ponadto, aktywność RNaz ulega podwyższeniu np. w okresie kiełkowania nasion czy w trakcie zachodzenia procesu apoptozy. Dodatkowo, niektórzy autorzy (np. Lehmann i wsp. 2001, czy Raj i wsp. 2001), donoszą, że do stymulacji aktywności Rnaz dochodzi także podczas ataku fitopatogenów i w warunkach niedoboru fosforu [3].
Udział RNazy P w powstawaniu tRNA
Wszystkie rodzaje tRNA powstają z dłuższych transkryptów tzw. pre-tRNA. Zanim cząsteczki staną się właściwym tRNA przechodzą proces dojrzewania, na który składa się kilka etapów:
RNaza P zbudowana jest z jednej cząsteczki RNA (tworzącej centrum ) i jednej cząsteczki białka (odpowiedzialnego za utrzymanie aktywnej konformacji). Jako enzym RNaza P zalicza jest do endonukleaz (podobnie jak RNazy D,E czy F) [4], [5]. Rybonukleaza P(RNaza P) jest więc kluczowym enzymem w biogenezie tRNA, który katalizuje endonukleolityczne rozszczepienie prekursorów tRNA oraz generuje ich dojrzały koniec 5’ [5]. Jedną z pierwszych katalitycznie scharakteryzowanych rybonukleaz była RNaza P pochodząca z bakterii [5].
Izolacja RNazy P z keratynocytów skóry (wg Pavlidou D. i wsp. 2005).
Jako materiał do badań wykorzystano keratynocyty ludzkiej skóry, odpowiednio przygotowane. Komórki pobrano ze skóry pacjentów z otyłością poddanych wcześniej abdominoplastyce (plastyka brzucha) . Komórki przygotowuje się przez odpowiednią preparatykę, na końcu próbki poddaje się wirowaniu w 4C przez 5 minut, a żywotnośc keratynocytó mierzy się testem z błękitem trypanu [6].
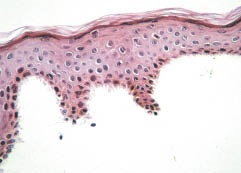
Fig. 1. Complete separation of human epidermis from dermis subsequent to incubation with dispase (haematoxylin-eosin, ×400). Zdjęcie: Kompletny podział ludzkiego naskórka od skóry właściwej po inkubacji z dispazą (naturalna proteaza), http://www.medicaljournals.se/acta/content/?doi=10.2340/00015555-0019&html=1
Oczyszczanie enzymu:
Uzyskane komórki w ilości 5x106 zawieszono w 4 ml lodowatego buforu A (tj. 10 mM HEPES o pH=7.9; 10 mM KCl; 0,1 mM EGTA; 1 mM ditriotreitol (DTT); 0,5 mM fluorek fenylometylosulfonylu (PMSF)) i pozostawiono do spęcznienia na 30 minut. Następnie, dodano (0,6% v/v)detergent Nonidet P-40(octyl phenoxylpolyethoxylethano), po czym zawiesinę homogenizowano, po czym odwirowano. Po wirowaniu otrzymane peletki zawieszono w 1 ml lodowatego buforu C (tj.: 20 mM HEPES o pH=7.9; 0,4 M NaCl; 1mM EDTA; 1mM EGTA; 1mM ditiotreitol; 20% gliceryna), próbkę energicznie wytrząsanow temperaturze 4st.C przez 30 minut. Zanieczyszczenia jądrowe usunięto przez 15 minutowe odwirowanie przy 14000 obr./minutę, po czym ekstrakt przechowywano w -20 st.C [6].
Komórkowe i jądrowe ekstrakty dializowano przez noc w 2 L buforu B (tj.: 50 mM fosforan potasu, pH=7.0; 0,1 mM ditiotreitol; 10% glicerol objetościowo), po czym próbkę w obj. 3 ml naniesiono na kolumnę fosfocelulozową (P-11) zrównoważoną wcześniej buforem B (jak wyżej). Kolumnę pzremywano tym samym buforem do momentu, aż nie zaobserwowano spadku absorbancji A=280 nm do zera. RNazę P eluowano z kolumny za pomocą 9 ml liniowego gradientu od 50 do 500 mM NH4Cl w buforze B. Otrzymane aktywne frakcje enzymu połączono i dializowano przez noc w 2 L buforu K (tj.: 5 mM MgCl2; 100 mM NH4Cl; 50 mM tris-HCl o pH=7.5) w obecności 20% glicerolu. Próbkę podzielono na małe porcje , po czym przechowywano w temperaturze -20 st.C [6].
Aktywność rybonukleolityczna
W homogenatach uzyskanych z komórek zwierzęcych występują dwa optymalne stężenia jonów wodorowych. W pH kwasowym mieszczącym się w granicach 5.2- 5.8 oraz pH zasadowym (7.8 – 8.2) stwierdza się optymalną aktywność RNaz. Co więcej, przeprowadzone badania wykazały, że aktywność tego enzymu w pH kwasowym jest mniejsza, niż w pH zasadowym. Między RNazami kwasowymi i zasadowymi występuje kilka różnić m.in. rybonukleazy kwasowe są wrażliwe na temperaturę i tracą swoją aktywność w rozcieńczonych kwasach, z kolei RNazy zasadowe są stosunkowo odporne na działanie wymienionych czynników [1].
Metoda Mc Donald- izolacja RNazy z tkanki trzustkowej
Metodę otrzymywania krystalicznej rybonukleazy po raz pierwszy opisał Kunitz (1940). Od tego czasu prowadzono badania, w trakcie których wykazano, że enzym ten nie tylko ma zdolność degradowania kwasu rybonukleinowego, ale także do hydrolizy białka [7]. Procedura opisana przez Kunitz’a została nieznacznie zmieniona na przestrzeni lat. W metodyce zmieniono kilka etapów, dzięki czemu udało się otrzymać RNazę wolną od wszelkich zanieczyszczeń, a cząsteczna RNazy jest praktycznie nienaruszona [7].
Metoda izolacji opiera się na ekstrakcji RNazy z tkanki za pomocą 0,12 M roztworu H2SO4. Kolejnym etapem jest frakcjonowanie za pomocą (NH4)2SO4. Całą preparatykę należy wykonywać w temperaturze 4st.C [1].
Otrzymywanie surowego preparatu rybonukleazy
Wykonanie:
Ekstrakcja I: Jako materiał do badań należy wykorzystać trzustkę bydlęcą, z której ekstrahuje się RNazę za pomocą lodowatego 0,12 M roztworu kwasu siarkowego (H2SO4). Cały proces prowadzis ię pzrez 24 godziny, po czym kwasowy ekstrakt pozostawia się w niskiej temperaturze, a trzustkę poddaje się oczyszczaniu (usunięcie tłuszczu oraz tkanki łącznej).
Kolejnym etapem jest homogenizacja tkanki, którą ponownie zalewa się zimnym roztworem H2SO4 (0,12 M) w proporcji: 2 L kwasu na 1 L zmielonej tkanki. Kolejną ekstrakcję prowadzi się przez 18 do 24 godzin, ze sporadycznym wytrząsaniem. Po ekstrakcji, homogenat trzustki należy przesączyć przez gazę, a pozostałą tkankę zalewa się równą objętością 0,12M H2SO4 . Tak przygotowaną próbkę pozostawia się na 1 godzinę [1], [7].
Przesącz otrzymany z trzech kolejnych ekstrakcji należy połączyć, po czym doprowadzić do 65% nasycenia roztworem (NH4)2SO4. W tym celu dodaje się 430 g (NH4)2SO4 w substancji na 1 litr przesączu. W celu uformowania osadu , otrzymany ekstrakt pozostawia się na 24 godziny, po czym odsącza wysolone białka przez sączki wykonane z bibuły Whatman nr 2 [1], [7].
Powstały przesącz zachowuje się, a osad traktuje się równą objętością wody (w stosunku do użytej ilości trzustki bydlęcej) wraz z dodatkiem 430 g (NH4)2SO4 na 1 litr zawiesiny. Tak przygotowaną próbkę ponownie pozostawia się na 24 godziny. Po upływie czasu inkubacji próbkę odsącza się (jak wyżej), a osad odrzuca [1], [7].
Wysolenie surowej frakcji enzymu:
W tym celu połączone przesącze doprowadza się do do 80% nasycenia (NH4)2SO4, dodając 105 gramów (NH4)2SO4 na 1 litr roztworu i odstawia na 24 godziny. Po wytrąceniu osadu ciecz należy ostrożnie zdekantować , a dalej odwirować przy 400 obr./minutę w wirówce z chłodzeniem. Otrzymany w ten sposób osad stanowi surowy preparat RNazy [1], [7].
Oczyszczanie RNazy
W celu oczyszczenia preparatu RNazy z enzymów proteolitycznych, należy wcześniej otrzymany osad enzymu rozpuścić w wodzie w proporcji 5 ml wody na 1 g osadu. Ponadto, do roztworu enzymu należy dodać 20% nasycony roztwór (NH4)2SO4 (pH=3) z wyliczeniem 20 ml roztworu (NH4)2SO4 na 1 g RNazy. Roztwór (NH4)2SO4 doprowadza się do odpowiedniego pH za pomocą 5M roztworu kwasu siarkowego.
Tak przygotowaną próbke podgrzewa się do temperatury w granicach 95 st.C - 100 st.C stale mieszając, po czym próbkę gwałtownie się oziębia do temperatury ok. 20-25 st.C. Następnie, poddaje się próbkę 1-godzinnej inkubacji (w tej temperaturze). W trakcie inkubacji dochodzi do wytrącenia się enzymó proteolitycznych, które następnie adsorbowane są na ziemi okrzemkowej (1 g ziemi okrzemkowej na 100 ml zawiesiny). Osad pzresącza sie na lejku Buchnera, a następnie przemywa się 3-krotnie za pomocą małych ilości zakwaszonego do pH=3 roztworu (NH4)2SO4 (20% nasycenie). W dalszym etapie oczyszczania, otrzymany przesącz doprowadza się do dp 50% nasycenia (NH4)2SO4 (dodając 18,8 g (NH4)2SO4 na 100 ml cieczy). Po upływie godzinnej inkubacji, osad należy odsączyć, po czym go odrzucić. RNazę odzyskuje się z przesączu po wysoleniu za pomocą siarczanu amonu o 80% nasyceniu ( w tym celu dodaje się 21 g (NH4)2SO4 na 100 ml przesączu). Po upływie 24 godzin należy odwirować osad enzymu [1], [7].
Krystalizacja jest metodą oczyszczania związków chemicznych. Cały proces polega na otrzymaniu nasyconego roztworu substancji, którą oczyszczamy w temperaturze wrzenia rozpuszczalnika. Następnie, roztwór poddaje się sączeniu (oddzielenie ewentualnych nierozpuszczalnych zanieczyszczeń) i ochłodzeniu (otrzymanego przesączu) [11].
Zazwyczaj, stałe związki organiczne, które są bezpośrednio wydzielone w określonej reakcji nie są chemicznie czyste (zawierają niewielkie ilości innych związków określanych mianem zanieczyszczeń). Zanieczyszczenia powstają jednocześnie z głównym produktem reakcji, w związku z czym próbki takie wymagają oczyszczenia. W większości przypadków oczyszcza się je przez krystalizację z odpowiednim rozpuszczalnikiem lub z mieszaniną rozpuszczalników.
Sam proces krystalizacji polega na wykorzystaniu różnicy rozpuszczalności związków w odpowiednich rozpuszczalnikach (lub ich mieszaninach) [12].
Do podstawowych etapów krystalizacji zalicza się:
Przygotowanie RNazy do krystalizacji
Otrzymany czysty preparat RNazy (oczyszczony z enzymów proteolitycznych) w ilości 1 g rozpuszcza się w 5 ml wody, po czym za pomocą 5M roztworu wodorotlenku sodu doprowadza się go do pH=4.8. Po uzyskaniu pożądanego pH do próbki dodaje się 5 ml nasyconego roztworu siarczanu amonu (na 1 g enzymu dodaje się 1 g ziemi okrzemkowej na 100 ml cieczy). Osad odsącza się i odrzuca, zaś przesącz doprowadza się do niższego pH=4.2 (0,5 M roztworem kwasu siarkowego – H2SO4) i wysala RNazę, doprowadzając do 70% nasycenia siarczanem amonu przez dodanie 67 ml nasyconego roztworu (NH4)2SO4 na 100 ml przesączu. Po upływie 24 godzin inkubacji w 4 st.C osad rybonukleazy należy odwirować przy 400 obr./minutę [1], [7].
Krystalizacja RNazy z siarczanem amonu
Całą preparatykę należy prowadzić w temperaturze 0- 4 st.C .
Enzym przygotowany do krystalizacji rozpuszcza się w 1 ml wody ( w stosunku 1g enzymu : 1 ml H2O). Występujące w próbce substancje zanieczyszczające adsorbuje się na ziemi okrzemkowej dodanej w stosunku: 5 g ziemi okrzemkowej na 100 ml roztworu. Powstały osad odsącza się na bibule, po czym przemywa się za pomocą wody.
Następnie, otrzymany przesącz należy doprowadzić do pH równego 4.2- w tym celu stosuje się 1 M roztwór wodorotlenku sodu (NaOH) lub 0,5 M roztwór kwasu siarkowego (H2SO4)- próbkę należy delikatnie mieszać, aż do momentu pojawienia się opalizującego zmętnienia roztworu. Proces krystalizacji należy przeprowadzać w cieplarce o temp. 20-25 st.C. Po upływie 3 dni, powstały osad odwirowuje się, a ług pokrystalizacyjny doprowadza się ponownie do pH równego 4.2 i znów krystalizuje (jak wyżej) [1], [7].
Proces rekrystalizacji
Krystaliczną RNazę rozpuszcza się wodzie, stosując proporcję: 2 ml wody na 1 g otrzymanego skrystalizowanego enzymu. Do rozpuszczonej próbki dodaje się 0,2 g ziemi okrzemkowej (na 2 ml roztworu), a całośc odsącza się na bibule. Pozostały po sączeniu osad (znajdujący się na sączku) należy przemyć małymi porcjami wody, a następnie doprowadzić do pH=4.2 (dodaje się nasyconego roztworu (NH4)2SO4 do momentu pojawienia się delikatnej opalescencji w próbce. Taką próbkę krystalizuje się ponownie (w temp. 20-25 st.C) prze 48 godzin. Otrzymany krystaliczny osad poddaje się wirowaniu, zaś otrzymany po wirowaniu supernatant doprowadza się ponownie do pH=4.2. Supernatant dalej krystalizuje się przez okres 3 dni (jak wyżej) [1], [7].
Rekrystalizowaną Rnaze można również rozpuścić w wodzie (w 1,5 ml) i dializować wobec wody. Cały proces należy prowadzić w temperaturze 0-4 st.C przez 24 godziny. Do dializatu w temp. 5 st.C , dodaje się następnie 96% roztwór etanolu (w stosunku: 12 ml etanolu na 1 ml roztworu). Dochodzi do formowania się osadu (w trakcie odstania próbki przez 2 dni w temperaturze 10 do 20 st.C). Odstany po czasie inkubacji krystaliczny osad, należy przemyć kilka razy 96% etanolem, po czym wysuszyć w eksykatorze nad CaCl2. Opisany proces nazywa się krystalizacją z etanolu [1], [7].
Onkonaza - białko z aktywnością rybonukleazy
Onkonaza (ONC) jest stsounkowo niedawno poznanym białkiem, które otrzymuje się z jaj żaby Rana pipiens. Jest białkiem o masie cząsteczkowej rónej 12000D, a buduje je pojedynczy łańcuch polipeptydowy składający się ze 104 aminokwasów. Onkonaza podobnie jak inne RNazy wykazuje wysoki stopień powinowactwa do niektórych RNaz występujących w organizmie człowieka (np. do RNazy trzustkowej czy wątrobowej)[8].
Przeprowadzone badania wykazały, że białko spełnia szereg bardzo ważnych funkcji. Działanie biologiczne tego białka, a zwłaszcza przeciwnowotworowe, jest ściśle związane z aktywnością rybonukleazy. Onkonaza prowadzi do śmierci komórki, przez rozkład wewnątrzkomórkowego RNA, ponadto odpowiada za hamowanie syntezy białek, a także wzrostu i proliferacji. Ponadto, wykazano, że odpowiada za selektywną indukcję apoptozy komórek nowotworowych (programowana śmierć). Ważnym elementem jej działania cytotoksycznego jest również aktywność antyoksydacyjna [8]. Podobnie jak inne RNazy, onkonaza ma zdolność rozkładania ds RNA (jednoniciowego RNA). Może również tworzyć aktywne biologicznie dimery.
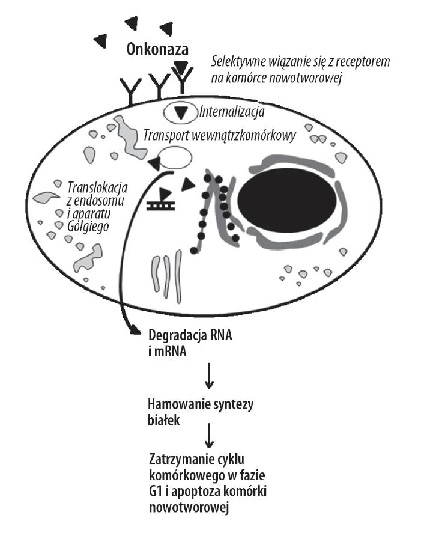
Zdjęcie: Mechanizm działania białka onkonazy [8]. http://www.phmd.pl/fulltxthtml.php?ICID=905228
Onkonaza uważana jest za najmniejsze białko rodziny RNaz A, które zostało wyizolowane z oocytów lub wczesnych embrionów żaby (Rana pipiens). Onkonaza jest białkiem stabilnym (temperatura denaturacji) nawet w temperaturze sięgającej 87 st.C, ponadto jest odporna na degradację przez różne proteazy. Białko to indukuje apoptozę komórek nowotworowych, a samo posiada niską cytotoksyczność [9].
W przeprowadzonych badaniach Saxena S. K. i wsp. (2003), stwierdzili, że onkonaza jest mniej wydajnym enzymem niż RNaza A. W optymalnych warunkach onkonaza wykazała się kilkaset razy niższą szybkością reakcji w porównaniu do RNazy A. Optymalnym pH działania dla onkonazy okazało sie pH=6.0, w którym to onkonaza wykazuje mniejszą różnicę szybkości reakcji w porównaniu do RNazy A [10].
Autor: Lidia Koperwas
Literatura:
[1]. Kłyszejko-Stefanowicz L, 2003. Ćwiczenia z biochemii. Wydawnictwo Naukowe PWN, 2003, s.544-548
[2]. Dolnicki A., 1979. Wpływa stymulatoró wzrostu na aktywność rybonukleaz i na metabolizm kwasów rybonukleinowych u roślin. Wiadomości botaniczne Tom XXIII - zeszyt 3, 1979.
[3]. Sytykiewicz H., Czerniewicz P., Leszczyński B., Sempruch C., Goławska S., Sprawka I., 2011. Aktywność rybonukleolityczna w liściach Czeremchy Zwyczajnej w okresie żerowania mszycy czeremchowo-zbożowej (Rhopalosipium Padi L.). Progress in Plant protection /Postępy w Ochronie Roślin 51 (1) 2011. http://www.progress.plantprotection.pl/pliki/2011/PPP_51_1_29_Sytykiewicz_H.pdf
[4]. Żak I. Kwasy nukleinowe, rozdział 19. http://biochigen.sum.edu.pl/podrecznik/19.pdf
[5]. Kudła M. Rybozymy. RNA- nośnik informacji i narzędzie katalizy enzymatycznej. http://www.igib.uw.edu.pl/~knbm/pdf/rybozymy.pdf
[6]. Pavlidou D., Vourekas A., Monastirli A., Kalavrizioti D., Tsambaos D., Drainas D., 2005. Isolation of Ribonuclease P Activity From Human Epidermis and its Regulation by Retinoids In vitro. Acta Derm Venereol 2006; 86: 114–118. http://www.medicaljournals.se/acta/content/?doi=10.2340/00015555-0019&html=1
[7]. McDonald M.R., 1948. A method for the preparation of “protease-free” crystalline ribonuclease. The Journal of General Physiology. http://www.ncbi.nlm.nih.gov/pmc/articles/PMC2213746/pdf/39.pdf
[8]. Zwolińska M., Smolewski P., 2010. Onkonaza-rybonukleaza o aktywności przeciwnowotworowej. Postepy Hig Med Dosw. (online), 2010; 64: 58-66. http://www.phmd.pl/fulltxthtml.php?ICID=905228 [9]. Meng Qiao, Li-Dong Zu, Xiao-Hong He, Ru-Ling Shen, Qing-Cheng Wang, ,Mo-Fang Liu , 2012. Onconase downregulates microRNA expression through targeting microRNA precursors. Cell Research (2012) 22:1199–1202. doi:10.1038/cr.2012.67; published online 24 April 2012. http://www.nature.com/cr/journal/v22/n7/full/cr201267a.html
[10]. Saxena S.K., Shogen K., Ardelt W., 2003.Alfacell Corporation, Bloomfield, NJ. Onconase and it’s therapeutic potential. Laboratorymedicine, may 2003. Number 5, volume 34. http://labmed.ascpjournals.org/content/34/5/380.full.pdf
[11]. http://www.e-biotechnologia.pl/Artykuly/krystalizacja
[12]. http://biomist.pl/chemia/artykuly/krystalizacja/43
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje