

|
Zamknij X
|
Uniwersytet Marii Curie-Skłodowskiej, Wydział Chemii, Zakład Radiochemii i Chemii Koloidów, Pl. M. Curie-Skłodowskiej 3, 20-031 Lublin, tel. (81) 5375622, k.szewczuk-karpisz@wp.pl
Streszczenie
Biosynteza białek to skomplikowany proces niezbędny do życia każdej komórce. Zachodzi on przy udziale rybosomów – organelli złożonych z białek oraz kwasów rybonukleinowych, a także wielu czynników translacyjnych. Informacja o każdym syntetyzowanym białku jest zawarta w kodzie genetycznym. Dokładne i wierne przepisanie sekwencji nukleotydowej DNA poprzez sekwencję mRNA na język aminokwasów jest niezbędne do prawidłowego funkcjonowania nowo syntetyzowanego białka. Ze względu na wielkie znaczenie procesu biosyntezy białek dla komórki maszyneria translacyjna jest często celem antybiotyków posiadających właściwości bakteriobójcze lub bakteriostatyczne.
W pracy dokonano przeglądu antybiotyków oddziałujących na wybrane miejsca aktywne rybosomów, tj. centrum dekodujące, centrum peptydylotransferazy oraz kanał wyjścia. Pełne zrozumienie mechanizmu działania opisanych substancji bakteriobójczych nie jest możliwe bez wcześniejszego poznania podstawowych zasad przepisywania informacji genetycznej, a także budowy rybosomów. Dlatego też, pracę tę uzupełniono o powyższe wiadomości.
W latach 50. XX wieku Francis Crick zaproponował kierunek przepisywania informacji genetycznej, tj. z DNA poprzez RNA na białka. W ten sposób powstał główny dogmat biologii molekularnej mówiący, że „z DNA powstają cząsteczki RNA, z których tworzą się białka” (TURNER i współaut. 2007). Jednakże, z biegiem lat stwierdzono, że istnieją pewne modyfikacje przedstawionego przepływu informacji genetycznej. Przykładowo, u retrowirusów (do których zaliczono wirus HIV) istnieje możliwość przepisania informacji z jednoniciowej cząsteczki RNA na dwuniciową cząsteczkę DNA. Jest to proces odwrotnej transkrypcji, zachodzący przy udziale enzymu określonego jako odwrotna transkryptaza (ZHENG i współaut. 2005). Ponadto, istnieje możliwość replikacji RNA m.in. u wirusów zapalenia wątroby typu C (BARTENSCHLAGER i LOHMANN, 2000). Podstawowy przepływ informacji genetycznej wraz z jego modyfikacjami przedstawiono na rysunku 1.
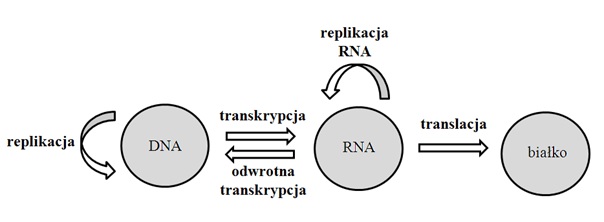
Rys. 1. Schemat dogmatu biologii molekularnej.
Replikacja to proces powielania materiału genetycznego, w który są zaangażowane specyficzne enzymy. W przypadku replikacji DNA proces zaczyna się od rozwinięcia dwuniciowej helisy w miejscu określanym jako widełki replikacyjne. Następnie do każdej nici zostaje dobudowana nowa komplementarna nić według reguły parowania zasad (tj. adenina z tyminą, cytozyna z guaniną). Synteza obu nici zachodzi jednocześnie, jednakże mechanizm ich tworzenia jest odmienny. Tzw. nić wiodąca jest syntetyzowana w sposób ciągły w kierunku od 5’ do 3’. Druga nić macierzysta określona jako nić opóźniona, jest tworzona w kierunku przeciwnym do miejsca inicjacji w postaci tzw. fragmentów Okazaki. Jej kopiowanie rozpoczyna się, gdy wskutek rozplatania dwuniciowej cząsteczki DNA powstaje odcinek o długości ok. 100-200 nukleotydów (Eucaryota). Po ukończeniu syntezy nici opóźnionej fragmenty Okazaki są łączone w jedną cząsteczkę DNA przy udziale ligazy DNA. (HAMES i HOOPER, 2006). Uproszczony schemat przedstawiający mechanizm replikacji przedstawiono na rysunku 2.
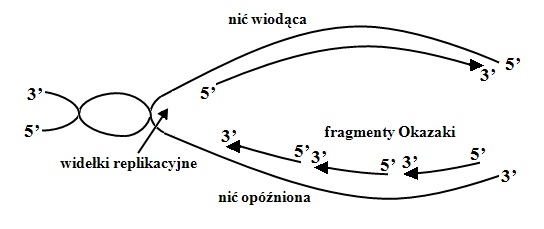
Rys. 2. Schemat replikacji DNA.
Proces przepisywania informacji genetycznej z cząsteczek DNA na cząsteczki RNA to transkrypcja. Podobnie jak replikacja oparta jest na regule parowania zasad, z tym że zamiast tyminy do cząsteczki RNA wbudowywany jest uracyl. Pojedyncza nić RNA tworzona jest w kierunku od 5’ do 3’ na postawie jednej z nici (tzw. nici sensownej) helisy DNA. Transkrypcja jest przeprowadzana przez polimerazę RNA, która wiąże się ze specyficznymi sekwencjami DNA w pobliżu końca 5’. W organizmach eukariotycznych występują trzy rodzaje tego enzymu, które przeprowadzają transkrypcję różnych genów. Enzymy te wykazują odmienną odporność na toksynę obecną w grzybach – α-amanitynę. W trakcie procesu transkrypcji helisa DNA ulega lokalnemu rozwinięciu. Terminacja procesu zachodzi przy udziale struktury typu „spinki do włosów” lub czynnika białkowego zwanego Rho (BROWN, 2001).
Ostatnim etapem przepływu informacji genetycznej jest proces translacji. Jest to wysoce skomplikowany proces syntezy białek, w który zaangażowane są m.in. organella komórkowe zwane rybosomami (KŁYSZEJKO-STEFANOWICZ, 2002).
Charakterystyka rybosomów
Rybosomy to rybonukleoproteinowe kompleksy obecne zarówno w komórce prokariotycznej, jak i eukariotycznej. Ich ilość jest skorelowana z aktywnością metaboliczną komórki i wynosi ok. 20 000 w komórce E. coli i ok. 100 000 w retykulocytach królika (KŁYSZEJKO-STEFANOWICZ, 2002). Rybosomy biorą czynny udział w procesie biosyntezy białka. Występują w cytoplazmie jako struktury wolne lub związane z siateczką śródplazmatyczną (BERG i współaut. 2007). Organella te po raz pierwszy zaobserwowano w mikroskopie świetlnym na początku XX wieku. Struktura i wielkość rybosomów zostały określone na podstawie wyników badań Svedberga (BROWN, 2001).
Każdy rybosom składa się z dwóch podjednostek określanych jako mniejsza i większa, różniących się ilością i rodzajem zawartych w nich białek oraz kwasów rybonukleinowych (rRNA). Białka rybosomalne umiejscowione są wokół rRNA i stabilizurą strukturę rybosomu. Natomiast rRNA stanowi jego szkielet i posiada bardzo rozwiniętą strukturę przestrzenną. Jest on zasadniczą częścią katalityczną rybosomu niezbędną w procesie biosyntezy białek (BERG i współaut., 2007).
Wykorzystanie techniki ultrawirowania przez Svedberga spowodowało, że wielkość rybosomów wyraża się poprzez stałą sedymentacji (S) (BROWN, 2001). Rybosomy prokariotyczne są mniejsze niż eukariotyczne. Ich masa wynosi 2,7 MDa, a stała sedymentacji – 70S (BERG i współaut. 2007).
Topologie rybosomu prokariotycznego i eukariotycznego są do siebie zbliżone, zwłaszcza w budowie podjednostek dużych. Zarówno podjednostka 50S E. coli, jak i 60S S. cerevisiae posiadają takie same wyniosłości, tj. wypukłość centralną (ang. central protuberance), kciuk rybosomalny (ang. stalk) oraz wyniosłość L1. Powierzchnia kontaktu z małą podjednostką w przypadku podjednostki 50S i 60S jest również bardzo zbliżona. Istotną różnicą wydaje się fakt, że struktura dużej podjednostki eukariotycznej jest rozciągnięta wzdłuż osi poprzecznej, co przyczynia się do jej elipsoidalnego kształtu, podczas gdy duża podjednostka prokariotyczna jest półkolista (GABASHVILI i współaut. 2000, GAO i współaut. 2003, FRANK 2000, VERSCHOOR i współaut. 1998).
W przypadku struktur małych podjednostek rybosomu Procaryota i Eucaryota różnice są bardziej widoczne. Podjednostka 30S ma kształt trapezoidalny, a w jej strukturze zaznaczają się charakterystyczne elementy: głowa (ang. head), korpus (ang. body), ramię (ang. shoulder), platforma (ang. platform), dziób (ang. beak) oraz ostroga (ang. spur). Pomiędzy głową i platformą znajduje się szczelina (ang. cleft) stanowiąca zasadniczą część katalityczną – tzw. centrum dekodujące rybosomu (GABASHVILI i współaut. 2000; BĄKOWSKA, 2005). W małej podjednostce eukariotycznej są obecne dodatkowe struktury. Są to tzw. stopy (ang. foot): prawa i lewa, znajdujące się w dolnej części korpusu (SPAHN i współaut. 2001). Warto również zauważyć, że widoczne jest znaczne powiększenie rozmiarów małej podjednostki Eucaryota w porównaniu z małą podjednostką Procaryota (VERSCHOOR i współaut. 1998). Topologię dwóch podjednostek rybosomu prokariotycznego przedstawiono schematycznie na rysunku 3.
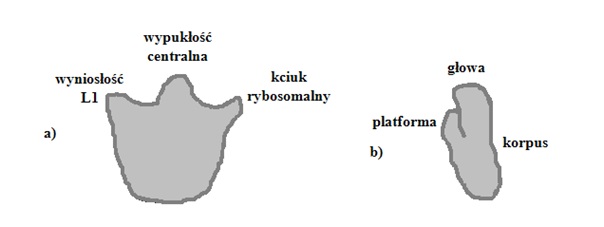
Rys. 3. Budowa rybosomu prokariotycznego: a) dużej podjednostki, b) małej podjednostki (na podstawie: TURNER i współaut. 2007).
Procedura biosyntezy białek
Główne etapy procesu biosyntezy białek – translacji, są uniwersalne i nie obserwuje się znacznych różnic w ich przebiegu w komórkach prokariotycznych i eukariotycznych. Proces ten jest tak fundamentalnym wydarzeniem w każdej żywej komórce, że niemożliwa jest wyraźna zmiana jego mechanizmu w toku ewolucji (KŁYSZEJKO-STEFANOWICZ, 2002).
Biosynteza białka zachodzi w dwóch podstawowych etapach. Pierwszy z nich to aktywacja aminokwasu, drugi – synteza łańcucha polipeptydowego z udziałem m.in. rybosomów. Proces aktywacji aminokwasów został opisany już w latach pięćdziesiątych (HOAGLAND i współaut. 1956), natomiast biosynteza białka do dziś nie została w pełni rozszyfrowana (KŁYSZEJKO-STEFANOWICZ, 2002). Aktywacja aminokwasu obejmuje dwa stadia, tj. (1) utworzenie aminoacyloadenylanu oraz (2) utworzenie aminoacylo-tRNA. Jest ona katalizowana przez specyficzną dla danego aminokwasu aminoacylo-tRNA syntetazę, określaną również jako ligaza. Końcowym rezultatem pierwszego etapu translacji jest wytworzenie cząsteczki tRNA związanej z resztą aminokwasową. Wiązanie pomiędzy grupą karboksylową tego aminokwasu i grupą hydroksylową rybozy tRNA ma charakter wiązania wysokoenergetycznego. W dalszych etapach translacji energia tego wiązania jest wykorzystywana podczas tworzenia wiązań peptydowych.
Synteza łańcucha białkowego jest wysoce skomplikowanym procesem angażującym szereg czynników translacyjnych. Obejmuje on trzy stadia, tj. (1) inicjację, (2) elongację i (3) terminację, które zachodzą cyklicznie. Początkowo następuje związanie mRNA z mniejszą podjednostką rybosomu, a następnie asocjacja obu podjednostek rybosomalnych. Każdy rybosom posiada trzy miejsca aktywne w swojej strukturze: miejsce P (dla peptydylo-tRNA), miejsce A (dla aminoacylo-tRNA) oraz miejsce E (dla tRNA pozbawionego reszty aminokwasowej). W czasie inicjacji do miejsca P przyłącza się tzw. inicjator, czyli cząsteczka tRNA połączona z metioniną. Natomiast podczas elongacji do miejsca A przyłącza się kolejne tRNA związane z odpowiednią resztą aminokwasową. Rodzaj aminoacylo-tRNA jest podyktowany kodonem mRNA, tj. specyficzną trójką nukleotydów. Wydłużanie łańcucha polipeptydowego odbywa się „o jeden aminokwas” zarówno u organizmów prokariotycznych, jak i eukariotycznych. A zatem, po utworzeniu wiązania peptydowego pomiędzy metioniną i aminokwasem dostarczonym przez tRNA do miejsca A, następuje przesunięcie ramki odczytu o jeden kodon mRNA (translokacja), wskutek czego tRNA pozbawione reszty aminokwasowej zostaje przesunięte w miejsce E i uwolnione, natomiast tRNA związane z dipeptydem zajmuje obecnie miejsce P. Miejsce A pozostaje wolne dla kolejnej cząsteczki tRNA niosącej wybraną resztę aminokwasową. Tworzenie wiązania peptydowego jest katalizowane przez peptydylotransferazę, która jest obecna w większej podjednostce rybosomu. Elongacja łańcucha polipeptydowego trwa do momentu osiągnięcia przez ramkę odczytu kodonu STOP. Wówczas następuje oddysocjowanie podjednostek rybosomalnych oraz uwolnienie nowosyntetyzowanego białka (KŁYSZEJKO-STEFANOWICZ, 2002; TURNER i współaut. 2007; BERG i współaut. 2007; BROWN, 2001).
Antybiotyki wpływające na aktywność rybosomu
Antybiotyki to substancje wykazujące działanie bakteriobójcze lub bakteriostatyczne nawet w bardzo niskich stężeniach. Pierwotnie określenia tego używano wyłącznie w odniesieniu do wtórnych metabolitów mikroorganizmów. Obecnie termin ten obejmuje również wszelkie półsyntetyczne i syntetyczne substancje posiadające takie same właściwości. Antybiotyki działają na ściśle określony element komórki bakteryjnej, np. ścianę komórkową, błonę cytoplazmatyczną, rybosomy lub szlaki metaboliczne. Ze względu na wiele różnic między komórką prokariotyczną i eukariotyczną istnieje małe prawdopodobieństwo toksycznego wpływu antybiotyku na organizm ludzki lub zwierzęcy. Ponadto, niektóre antybiotyki wykazują toksyczność selektywną, która pozwala na skuteczną eliminację patogenu bez szkody dla gospodarza (SINGLETON, 2000). Pod względem chemicznym antybiotyki są mocno zróżnicowaną grupą, co stanowi problem podczas ich klasyfikacji (MARKIEWICZ i KWIATKOWSKI, 2008).
Hamowanie biosyntezy białek, jako procesu niezbędnego do życia komórki, to jeden z mechanizmów działania antybiotyków. Poszczególne substancje mogą wpływać hamująco na wszystkie etapy translacji, oddziałując na rybosomy lub czynniki translacyjne (HARMS i współaut. 2003). Ze względu na wysoką skuteczność antybiotyki obniżające aktywność rybosomów są obiektem wielu badań biochemicznych, co skutkuje pojawieniem się licznych nowych generacji antybiotyków działających wydajniej i mniej toksycznie na organizm gospodarza (YONATH, 2005).
Antybiotyki działające na centrum dekodujące rybosomu
Centrum dekodujące to miejsce oddziaływania kodonu mRNA z komplementarnym anty-kodonem tRNA oraz tłumaczenia informacji genetycznej na język aminokwasów. Jest ono zlokalizowane w obrębie małej podjednostki rybosomu w pobliżu 3’ końca 16S rRNA (LYNCH i PUGLISI, 2001).
Antybiotyki aminoglikozydowe, do których zalicza się m.in. streptomycynę, higromycynę B, paromomycynę i neomycynę, oddziałują na małą podjednostkę, a ich miejscem docelowym jest właśnie centrum dekodujące lub rejony rRNA bezpośrednio z nim sąsiadujące. Związanie aminoglikozydów z centrum dekodującym powoduje zwykle zmianę konformacji rRNA prowadzącą do zakłóceń przepisywania informacji genetycznej, głównie do obniżenia wierności dekodowania (MEHTA i CHAMPNEY, 2002). Wszystkie antybiotyki z tej grupy wykazują powinowactwo wyłącznie do komórek bakteryjnych (MCCOY i współaut. 2010), zwłaszcza tlenowych pałeczek gramujemnych. Aminoglikozydy mają zdolność do swobodnej dyfuzji przez kanały w zewnętrznej błonie bakterii, jednakże do przekroczenia błony cytoplazmatycznej wymagają systemu transportu czynnego (MARKIEWICZ i KWIATKOWSKI, 2008). Istnieje możliwość uodpornienia się mikroorganizmów na antybiotyki aminoglikozydowe. Jest to następstwo enzymatycznych modyfikacji antybiotyku, tj. O-adenylacji, O-fosforylacji lub N-acetylacji, które zmniejszają powinowactwo antybiotyku do rRNA. Innymi sposobami nabywania odporności bakterii jest modyfikacja własnego rRNA, mutacja białek rybosomalnych, a także wykształcenie mechanizmu czynnie usuwającego cząsteczki antybiotyku z komórki (MCCOY i współaut. 2010).
Higromycyna B ogranicza ruchliwość helisy 44 biorącej udział w translokacji, co prowadzi do zatrzymania aminoacylo-tRNA w miejscu A (YONATH, 2005). Z kolei, streptomycyna zmienia konformację podjednostki 30S na taką, która wykazuje większe powinowactwo do niekomplementarnego tRNA, co prowadzi do syntezy białek o zmienionych właściwościach (HARMS i współaut. 2003). Paromomycyna i neomycyna oddziałują z wewnętrzną pętlą 16S rRNA, obecną wewnątrz centrum dekodującego. Ich związanie w ww. regionie powoduje zmiany konformacyjne pętli w miejscu A, które zwiększają jej powinowactwo do I i II pierścienia aminoglikozydów (MEHTA i CHAMPNEY, 2002).
Tetracyklina to antybiotyk będący wtórnym metabolitem niektórych organizmów, którego działanie opiera się na wiązaniu z podjednostką 30S i ograniczeniu ruchu aminoacylo-tRNA. W rezultacie cząsteczka ta nie może jednocześnie oddziaływać z miejscem dekodującym oraz centrum peptydylotrasferazy zlokalizowanym na dużej podjednostce 50S (MARKIEWICZ i KWIATKOWSKI, 2008). Tetracyklina wiąże się najczęściej pomiędzy głową i korpusem małej podjednostki, czyli w miejscu A.
Oprócz aminoglikozydów czy tetracykliny antybiotykami hamującymi biosyntezę białek poprzez oddziaływanie na centrum dekodujące są również spektynomycyna, paktamycyna oraz edeina. Pierwsza z nich należy do grupy aminocytoli i oddziałuje z głową małej podjednostki, hamując translokację peptydylo-tRNA z miejsca A do miejsca P (HARMS i współaut. 2003). Paktamycyna wiążąc się do małej podjednostki powyżej platformy blokuje ruch mRNA względem podjednostki 30S (BRODERSEN i współaut. 2000). Podobny mechanizm działania wykazuje edeina (YONATH, 2005).
Antybiotyki działające na centrum peptydylotransferazy
Centrum peptydylotransferazy (PTC) stanowi region dużej podjednostk, który kontaktuje się z trzema funkcjonalnymi miejscami rybosomu: aminoacylowym (miejsce A), peptydylowym (miejsce P) oraz usuwającym deacylowane tRNA (miejsce E) (BASHAN i współaut. 2003). PTC uczestniczy w tworzeniu wiązania peptydowego między grupami aminowymi i karboksylowymi aminokwasów poprzez odpowiednie w czasie i przestrzeni rozmieszczenie aminoacylo-tRNA i peptydylo-tRNA (BROWN, 2001).
Wiele mikroorganizmów produkuje substancje o charakterze antybiotyków, które zakłócają funkcjonowanie PTC. Mechanizm działania tych antybiotyków to zapobieganie tworzeniu wiązania peptydowego poprzez upodabnianie się do substratu, a także blokowanie tunelu dla syntetyzowanego łańcucha polipeptydowego (HARMS i współaut. 2003). Uniemożliwienie związania substratu do PTC jest najprostszym mechanizmem działania antybiotyków (YONATH, 2005). Do ww. grupy antybiotyków zalicza się m.in. chloramfenikol, linkozamidy, pleuromulityny, streptograminy, oksazolidinony oraz sparsomycynę (MCCOY i współaut. 2010).
Chloramfenikol, pierwszy naturalny antybiotyk wyprodukowany syntetycznie, wiążę się do podjednostki 50S w pobliżu centrum peptydylotransferazy, blokując przyłączanie aminoacylo-tRNA do miejsca A, co uniemożliwia tworzenie wiązania peptydowego. Aktywność chloramfenikolu jest optymalna po wysyceniu rybosomów antybiotykiem w stosunku 1:1. Dzięki prostej budowie jego cząsteczki łatwo pokonuje barierę osłon komórkowych (MARKIEWICZ i KWIATKOWSKI, 2008).
Klindamycyna, jako główny przedstawiciel linkozamidów, wiąże się do dużej podjednostki pomiędzy PTC a rybosomalnym tunelem wyjścia nowosyntetyzowanego polipeptydu, redukując elastyczność rybosomu w tym regionie. Ten unikalny sposób wiązania nadaje klindamycynie szczególną efektywność w zwalczaniu infekcji spowodowanych przez beztlenowe bakterie i Toxoplasma gonidii, a także w leczeniu pneumocystozowego zapalenia płuc wywołanego przez Pneumocystis carinii u osób cierpiących na AIDS (MEHTA i CHAMPNEY, 2002). Oprócz hamowania reakcji katalizowanej przez peptydylotransferazę, klindamycyna może również powodować oddysocjowanie peptydylo-tRNA od rybosomu (MCCOY i współaut. 2010).
Tiamulina z grupy antybiotyków pleuromulitynowych, wiąże się ściśle do dużej podjednostki w miejscu aminoacylowym, obejmując częściowo również miejsce peptydylowe. W ten sposób blokuje dwa ww. miejsca jednocześnie (MCCOY i współaut. 2010).
Streptograminy zostały podzielone na dwie grupy różniące się budową i mechanizmem działania. Streptograminy A to makrolaktony inaktywujące miejsce donorowe i akceptorowe peptydylotransferazy, a tym samym hamujące elongację. Streptograminy B to cykliczne heksapeptydy zaburzające właściwe ustawienie peptydylo-tRNA w miejscu P, co hamuje tworzenie wiązań peptydowych. W ten sposób streptograminy zakłócają zarówno wczesne, jak i późne etapy biosyntezy białka (MARKIEWICZ i KWIATKOWSKI, 2008; MCCOY i współaut. 2010). Działanie synergistyczne obu grup polega na tym, że streptograminy A powodują zmiany konformacji centrum peptydylotransferazy, co prowadzi do zwiększenia powinowactwa rybosomu do streptogramin B (HARMS i współaut. 2003).
Oksazolidinony to nowa klasa antybiotyków aktywna wobec bakterii gram dodatnich i prątków, do której zalicza się linezolid i eperezolid (HARMS i współaut. 2003). Ww. substancje konkurują z substratem o miejsce akceptorowe podjednostki 50S. Dodatkowo, linezolid zwiększa częstotliwość zmiany ramki odczytu oraz nonsensownej supresji translacji (LYNCH i PUGLISI, 2001). Oporność bakterii na linezolid pojawia się niezwykle rzadko (BASHAN i współaut. 2003).
Sparsomycyna oddziałuje zarówno na rybosomy prokariotyczne, jak i eukariotyczne. Do jej związania niezbędne jest tRNA obecne w miejscu peptydylowym dużej podjednostki (HANSEN i współaut. 2003). Przyłączenie sparsomycyny powoduje zmiany konformacyjne, które wpływają na umiejscowienie cząsteczek tRNA w obrębie rybosomu, a także stymuluje układ do spontanicznej translokacji prowadzącej do upośledzenia funkcjonowania rybosomu (MCCOY i współaut. 2010).
Antybiotyki działające na kanał wyjścia
Nowosyntetyzowany łańcuch polipeptydowy przemieszcza się przez tzw. tunel wyjścia położony poniżej centrum peptydylotransferazy. Antybiotyki wiążące się do tego regionu, do których zalicza się m.in. makrolidy, utrudniają przemieszczanie się polipeptydu przez tunel (MCCOY i współaut. 2010).
Makrolidy to rodzina makrocyklicznych laktonów zawierających zazwyczaj jeden lub więcej deoksymonosacharydów. Jednym z najbardziej znanych przedstawicieli makrolidów jest erytromycyna. Antybiotyki te wiążą się do węzła regionu wyjścia blokując przesuwanie łańcucha polipeptydowego. W rezultacie następuje zatrzymanie elongacji oraz odłączenie krótkich peptydylo-tRNA od rybosomu. Makrolidy oddziałują wyłącznie na rybosomy prokariotyczne. Czynnikiem odpowiedzialnym za ich selektywność jest guanina w pozycji 2058 rRNA u Eucaryota, zastąpiona u Procaryota przez adeninę. Częstym mechanizmem nabywania oporności na makrolidy i ketolidy jest upodabnianie się bakterii do organizmów eukariotycznych wskutek modyfikacji miejsca 2058 (MCCOY i współaut. 2010). Metylacja tej pozycji wywołuje oporność wyłącznie na makrolidy. Natomiast dimetylacja ww. miejsca chroni bakterie przed działaniem zarówno makrolidów, jak i ketolidów (LIU i DOUTHWAITE, 2002).
Podsumowanie
Dogmat biologii molekularnej opisuje kierunek przepływu informacji genetycznej od DNA do białka. Transkrypcja to proces przepisywania sekwencji nukleotydowej DNA na sekwencję nukleotydową mRNA. Z kolei, translacja jest syntezą łańcucha polipeptydowego na podstawie informacji zawartych w mRNA. W procesie biosyntezy białek zaangażowane są m.in. rybosomy – kompleksy białek i kwasów rybonukleinowych. Ich miejsca funkcyjne, pełniące kluczowe role podczas tworzenia polipeptydów, są częstym celem antybiotyków. Duża ilość dostępnych substancji bakteriobójczych i bakteriostatycznych, których mechanizm działania opiera się na zakłócaniu procesu biosyntezy białek, pozwala na skuteczną eliminację wybranych patogenów.
LITERATURA:
BARTENSCHLAGER R., LOHMANN V., 2000. Replication of hepatits C virus. J. Gen. Virol. 81, 1631-1648.
BASHAN A., AGMON I., ZARIVACH R., SCHULEZEN F., HARMS J., BERISIO R., BARTELS H., FRANCESCHI F., AUERBACH T., HANSEN H. A. S., KOSSOY E., KESSLER M., YONATH A., 2003. Structural basis of the ribosomal machinery for peptide bond formation, translocation and nascent chain progression. Molecular Cell 11, 91-102.
BĄKOWSKA K., 2005. Funkcja wybranych fragmentów 16S rRNA małej podjednostki rybosomalnej w procesie biosyntezy białka. Biotechnologia 2, 206-214.
BERG J. M., STRYER L., TYMOCZKO J. L., 2007. Biochemia, wydanie III. PWN, Warszawa.
BRODERSEN D. E., CLEMONS W. M., CARTER A. P., MORGAN-WARREN R. J., WIMBERLY B. T., RAMAKRISHNAN V., 2000. The scructural basis for the action of the antibiotics tetracycyline, pactamycin and hygromycin B on the 30S ribosomal subunit. Cell 103, 1143-1154.
BROWN T. A., 2001. Genomy. PWN, Warszawa.
GABASHVILI I. S., AGARWAL R. K., SPAHN C. M. T., GRASSUCCI R. A., SVERGUN D. I., FRANK J., PENCZEK P., 2000. Solution structure of the E. coli 70S ribosome at 11.5  resolution. Cell 100, 537-549.
resolution. Cell 100, 537-549.
GAO H., SENGUPTA J., VELLE M., KOROSTELEV A., ESWAR N., STAGG S. M., VAN ROEY P., AGARWAL R. K., HARVEY S. C., SALI A., CHAPMAN M. S., FRANK J., 2003. Study of the structural dynamics of the E. coli 70S ribosome using real-space refinement. Cell 113, 789-801.
HAMES B. D., HOOPER N. M., 2006. Biochemia – krótkie wykłady. Wydanie drugie. PWN, Warszawa.
HANSEN J. L., MOORE P. B., STEITZ T. A., 2003. Structures of five antibiotics bound at the peptidyl transferase center of the large ribosomal subunit. J. Mol. Biol. 333, 1061-1075.
HARMS J. M., BARTELS H., SCHLUZEN F., YONATH A., 2003. Antibiotics acting on the translational machinery. J. Cell Sci. 116, 1391-1393.
HOAGLAND M. B., KELLER E. B., ZAMECNIK P. C., 1956. Enzymatic carboxyl activation of amino acids. J. Biol. Chem. 218, 345-358.
KŁYSZEJKO-STEFANOWICZ L., 2002. Cytobiochemia. Biochemia niektórych struktur komórkowych. PWN, Warszawa.
LIU M., DOUTHWAITE S., 2002. Activity of ketolide telithromycin is refractory to erm monomethylation of bacterial rRNA. Antimicrobial Agents and Chemiotherapy, 1629-1633.
LYNCH S. R., PUGLISI J. D., 2001. Structure of a eucaryotic decoding region A-site RNA. J. Mol. Biol. 306, 1023-1035.
MARKIEWICZ Z., KWIATKOWSKI Z. A., 2008. Bakterie, antybiotyki, lekooporność. PWN, Warszawa.
MCCOY L. S., XIE Y., TOR Y., 2011. Antibiotics that target protein synthesis. Villey Interdiscip. Rev. RNA 2, 209-232.
MEHTA R., CHAMPNEY W. S., 2002. 30S ribosomal subunit assembly is a target for inhibition by aminoglicosides in Escherichia coli. Antimicrobial Agents and Chemiotherapy, 1546-1549.
SINGLETON P., 2000. Bakterie w biologii, biotechnologii i medycynie. PWN, Warszawa.
SPAHN C. M. T., BECKMANN R., ESWAR N., PENCZEK P. A., SALI A., BLOBEL G., FRANK J., 2001. Structure of the 80S ribosome from Saccharomyces cerevisiae – tRNA-ribosome and subunit-subunit interactions. Cell 107, 373-386.
TURNER P. C., McLENNAN A. G., BATES A. D., WHITE M. R. H., 2007. Biologia molekularna – krótkie wykłady. Wydanie drugie. PWN, Warszawa.
VERSCHOOR A., WERNER J. R., SRIVASTAVAS., ROBERT A., GRASSUCCI R. A., FRANK J., 1998. Three-dimensional structure of the yeast ribosome. Nucleic Acid Research 26, 655-661.
YONATH A., 2005. Antibiotics targeting ribosomes: resistance, selectivity, synergism and cellular regulation. Annu. Rev. Biochem. 74, 649-679.
ZHENG Y. H., LOVSIN N., PETERLIN B. M., 2005. Nowly identified host factors modulate HIV replication. Immunol. Lett. 97, 225-234.
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje