

|
Zamknij X
|
Horyzontalny transfer genów odgrywa główną rolę w ewolucji bakterii. Prowadzi on do ogromnej zmienności genetycznej mikroorganizmów w obrębie jednego gatunku. Horyzontalny transfer genów polega na stabilnym przeniesieniu informacji genetycznej z jednego organizmu do drugiego. U bakterii odbywa się on na drodze koniugacji, transdukcji lub transformacji [1].
Horyzontalny transfer genów
Horyzontalny transfer genów (ang. Horizontal Gene Transfer, HGT) zwany także transferem lateralnym (ang. Lateral Gene Transfer, LTG) polega na przeniesieniu materiału genetycznego z jednego organizmu do drugiego, gdzie przekazane geny podlegają utrwaleniu w genomie. Koncepcje horyzontalnego transferu genów jako międzygatunkowego przepływu genów wprowadził Michael Syvanen w 1984 roku. Międzykomórkowy transfer informacji genetycznej umożliwia bakteriom wzbogacenie własnej puli genów o dodatkową informację genetyczną pochodzącą często od niespokrewnionych taksonomicznie gatunków bakterii. Prowadzi to do ogromnej zmienności genetycznej mikroorganizmów w obrębie jednego gatunku i zapewnia im szybkie tempo ewolucji [1].
Wyróżniamy trzy podstawowe rodzaje horyzontalnego transferu genów: koniugacje, transdukcje oraz transformacje. Koniugacja polega na przekazywaniu genów poprzez bezpośredni kontakt komórek bakteryjnych. W procesie transdukcji wprowadzenie nowego genu do komórki bakteryjnej następuje pośrednio przez bakteriofagi. Natomiast transformacja polega na pobraniu przez bakterie natywnego DNA ze środowiska. Procesy koniugacji i transformacji wymagają bliskiego kontaktu drobnoustrojów, natomiast proces transdukcji umożliwia pozyskanie nowych genów z odległych niszy ekologicznych. Cechy nabywane na drodze horyzontalnego transferu genów umożliwiają bakteriom zasiedlanie nowych środowisk i często prowadzą do selekcji nowych wirulentnych szczepów bakteryjnych [1].
Koniugacja
Koniugacja została po raz pierwszy opisana w 1946 roku przez amerykańskiego genetyka Joshuę Lederberga. Polega ona na jednokierunkowym transferze materiału genetycznego z jednej komórki bakteryjnej (dawcy) do drugiej (biorcy). W większości przypadków koniugacja dotyczy plazmidowego DNA, choć u niektórych mikroorganizmów w ten sposób może być także przekazywane chromosomalne DNA. Bakteryjne systemy koniugacyjne uwarunkowane są obecnością w komórkach dawców plazmidów koniugacyjnych [1].
Plazmidy są to pozachromosomowe replikony (Rys.1). Ich wspólną właściwością jest zdolność do autonomicznej replikacji niezależnej od chromosomu bakteryjnego. Struktura plazmidu obejmuje replikon minimalny oraz replikon podstawowy. Replikon minimalny stanowią geny niezbędne w procesie replikacji, natomiast replikon podstawowy skupia geny odpowiedzialne za regulacje liczby kopii danego plazmidu, jego stabilność i zgodność z innymi plazmidami. Geny replikonu minimalnego kodują tylko niektóre enzymy konieczne w procesie replikacji, natomiast pozostałe, niezbędne w tym procesie enzymy dostarczane są przez komórkę bakteryjną. Poza informacją genetyczną warunkującą replikację na plazmidach, znajdują się geny, które pomimo, że nie mają wpływu na podstawowy metabolizm bakterii, mogą jej nadawać dodatkowe cechy fenotypowe takie jak: geny oporności na antybiotyki, geny kodujące toksyny, inwazyny i adhezyny, geny odpowiedzialne za produkcję kolicyn lub umożliwiające rozkład i asymilację różnych związków organicznych. Plazmidy wykazują różne właściwości i nadają komórkom bakteryjnym swoiste cechy, nie są one jednak konieczne do wzrostu bakterii w normalnych warunkach, ponieważ wszystkie geny metabolizmu podstawowego zlokalizowane są w chromosomie [1].
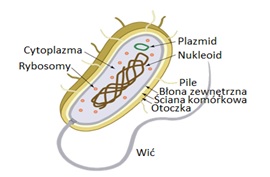
Rys.1. Budowa komórki bakteryjnej.
Większość plazmidów to kowalencyjnie zamknięte, koliste cząsteczki dwuniciowego DNA tworzące strukturę tzw. CCC-DNA. Jednak ostatnio coraz częściej izolowane są także plazmidy liniowe, które mogą występować w dwóch typach strukturalnych (Rys.2). Pierwszy z nich, jest to „prawdziwa” liniowa cząsteczka dwuniciogo DNA, posiadająca wolne końce. Cechą charakterystyczną tej struktury jest obecność białek terminalych (TP) połączonych z końcem 5’-łańcucha DNA. Białka te chronią DNA przed atakiem komórkowych 5’-egzonukleaz. Drugi typ strukturalny, nazwany typem „szpilki do włosów” (ang. hairpin loop), to liniowe cząsteczki dwuniciowego DNA, których końce są kowalencyjnie połączone. Plazmid tego typu to jeden polinukleotydowy łańcuch, który jest komplementarny na całej swej długości, za wyjątkiem krótkich naprzeciwległych odcinków, w wyniku czego powstają jednoniciowe pętle na końcach liniowej dwuniciowej struktury. Wielkość plazmidów jest bardzo zróżnicowana i zawiera się w granicach od około 1000 par zasad (1kb) do 1700 kb. Dolna wartość wielkości plazmidu wyznaczana jest minimalną ilością informacji genetycznej niezbędnej do kodowania funkcji potrzebnych do jego replikacji, nie pozostawiając miejsca na kodowanie żadnych innych cech fenotypowych. Natomiast górna granica wielkości plazmidu może nawet przekraczać wielkość chromosomów niektórych bakterii. Najczęściej duże plazmidy niosą geny warunkujące złożone procesy fizjologiczne jak np. zdolność do koniugacji, szlaki metaboliczne lub oporność na antybiotyki i inne substancje antybakteryjne. Znając wielkość plazmidu można określić teoretyczną pojemność kodującą plazmidu, czyli oszacować ile średniej wielkości białek może być kodowanych przez jego genom. Daje to możliwość określenia jaki procent całkowitej, niesionej przez komórkę informacji genetycznej, „przypada” na informację niesioną przez plazmid. Cechą charakterystyczną każdego plazmidu jest liczba kopii jego cząsteczek przypadająca na komórkę gospodarza. Wyróżniamy plazmidy jednokopiowe, kiedy to na komórkę przypada jedna cząsteczka plazmidu, niskokopiowe, gdy liczba kopii plazmidu wynosi 3-8 cząsteczek lub wysokokopiowe, dla których typowa liczba kopii wynosi 15-20. Ogólna zasada mówi, że im plazmid jest większy, tym liczba jego kopii jest mniejsza. Określony plazmid charakteryzuje się typowym dla siebie zakresem gospodarza. Plazmid o wąskim zakresie gospodarza funkcjonuje tylko w swoim naturalnym gospodarzu lub filogenetycznie zbliżonym. Natomiast plazmidy o szerokim zakresie gospodarza mogą funkcjonować w różnych, czasem bardzo odległych filogenetycznie gatunkach [2].
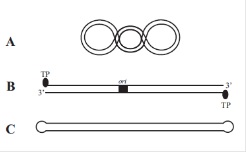
Rys.2. Formy strukturalne naturalnych plazmidów bakteryjnych. A - Plazmid kolisty w formie CCC-DNA. B – Plazmid liniowy. C – Plazmid liniowy typu „hairpin loop” [2].
Plazmidy koniugacyjne zawierają determinanty genetyczne, które umożliwiają proces koniugacji u bakterii. Zazwyczaj są to cząsteczki o wielkości około 30 kpz i dużej liczbie genów. Plazmidy koniugacyjne posiadają geny kodujące białka Tra oraz gen mob. Geny znajdujące się w regionie tra odpowiadają za utworzenie połączenia w postaci mostka koniugacyjnego przez który następuje transfer DNA. Po wytworzeniu mostka koniugacyjnego, enzym relaksaza, kodowany przez gen mob, nacina cząsteczkę DNA w miejscu określanym jako origin koniugacyjny (oriT, ang. origin of transfer) [1]. Gdy plazmid posiada komplet genów niezbedny do wytworzenia kontaktu z komórką biorcy i przekazania do niej DNA, nazywamy go plazmidem koniugacyjnym (Tra+).Znane są także plazmidy, zwane mobilizowalnymi (Tra– Mob+), które nie są zdolne do samoistnego przenoszenia informacji genetycznej, gdyż nie posiadają genów odpowiedzialnych za utworzenie połączenia koniugacyjnego. Zawierają za to geny kodujące białka przecinające plazmid w miejscu oriT. Plazmidy mobilizowalne mogą przekazać swój DNA do komórki biorcy jedynie przy współudziale dodatkowego plazmidu mobilizującego. Zdolność koniugacyjną plazmidu można określić poprzez odnalezienie w jego genomie sekwencji homologicznych z elementami innych systemów koniugacyjnych [2].
W komórkach Escherichia coli prototypem takiego plazmidu koniugacyjnego jest plazmid F. Może on występować w formie autonomicznej (komórki typu F) lub w formie zintegrowanej z chromosomem (komórki typu Hfr). Wyróżniamy trzy typy płciowe bakterii: F-, F+ i Hfr. O płci bakterii decyduje plazmid nazwany czynnikiem F. Komórki posiadające ten plazmid mają zdolność koniugowania z komórkami F- oraz przekazywania im części własnego DNA. Czynnik F może być zawarty w cytoplazmie komórek F+ lub zostać włączony do chromosomu bakterii w przypadku komórek Hfr. Komórki F+ oraz Hfr zawierające czynnik F nazywamy męskimi. Natomiast komórki F-nazywamy żeńskimi. Pomiędzy bakteriami typów F+ i F- oraz Hfr i F- może dochodzić do koniugacji [3].
W przypadku układu modelowego Escherichia coli/plazmid F koniugacja jest procesem wieloetapowym (Rys.3). Rozpoczyna się od kontaktu czubka pilusa z powierzchnią komórki biorcy. Pilus przesuwa się po powierzchni aż napotka właściwy receptor, którym jest białko błony zewnętrznej OmpA. Wówczas zostaje unieruchomiony i ulega retrakcji oraz degradacji, co prowadzi do kontaktu osłon komórkowych. Następnie dochodzi do ścisłego połączenia błon komórkowych. Pary tak złączonych komórek są stabilne i gotowe do przekazania DNA. Koniugacyjny transfer DNA rozpoczyna się od jednoniciowego nacięcia w specyficznym miejscu plazmidu F, zwanym oriT, przez białko TraI.Plazmidowo kodowana helikaza rozkręca podwójną helisę DNA, co umożliwia rozdział obu nici i przeniesienie jednej z nich do komórki biorcy przez mostek koniugacyjny. Podczas przekazywania do obu nici zostają dobudowane komplementarne nici DNA, które następnie ulęgają cyrkularyzacji. Synteza nici komplementarnych przebiega w komórce dawcy w sposób ciągły, natomiast w komórce biorcy przez fragmenty Okazaki. Komórka biorcy otrzymuje w ten sposób nie tylko dodatkową informację genetyczną, kodującą nowe cechy fenotypowe, ale także zdolność przekazywania kopii nowo uzyskanego plazmidu innym bakteriom [3].
System koniugacyjny zależny od plazmidu F występuje jedynie u Escherichia coli i innych pałeczek jelitowych. Plazmidy koniugacyjne zostały również wykryte u bakterii gramdodatnich. Jednak tutaj proces koniugacji może przebiegać według innego mechanizmu. W przypadku Enrerococcus faecalis plazmid koniugacyjny jest elementem składowym systemu odpowiadającego na produkowane przez komórkę biorcy feromony płciowe. Warunkuje on w komórce dawcy syntezę substancji agregacyjnej (adhezyny), która jest konieczna do utworzenia par lub agregatów koniugacyjnych [3].
Proces koniugacji warunkowany plazmidami o szerokim zakresie gospodarza odgrywa bardzo ważną rolę w horyzontalnym transferze genów w środowiskach naturalnych, co w długich przedziałach czasowych ma istotną rolę w procesie ewolucji. Koniugacja umożliwia przenoszenie genów plazmidowych pomiędzy bakteriami niekoniecznie należącymi do tego samego gatunku, co stanowi ważny sposób rozprzestrzeniania się informacji genetycznej pomiędzy różnymi, niespokrewnionymi taksonomicznie gatunkami bakterii. Plazmidy nadają bakteriom cechy selekcyjnie korzystne jak np. oporność na antybiotyki, chemioterapeutyki, sole metali czy zdolność do produkcji bakteriocyn. Przyczyniają się one także do efektywnego rozprzestrzeniania informacji genetycznej. Przykład może stanowić szybkie rozprzestrzenianie się bakterii opornych na leki np. Shigella oporna na różne antybiotyki czy gronkowiec oporny na penicylinę [3].
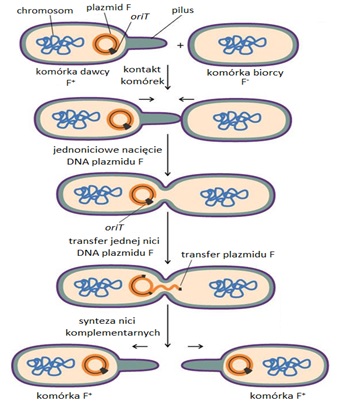
Rys.3. Proces koniugacji u bakterii [4].
Transdukcja
Transdukcja została odkrytaw 1952 roku przez Joshua Lederberga i Nortona Zindera w procesie infekcji komórek bakteryjnych bakteriofagami. Polega ona na przeniesieniu materiału genetycznego od komórki dawcy do biorcy przy udziale faga.Bakteriofag, infekując komórkę bakteryjną, może wbudować we własny genom odcinek DNA swojego gospodarza. Kiedy następnie ten sam wirus zakazi inną komórkę bakterii, może przekazać jej fragment DNA dawcy. Komórka biorcy zyskuje wówczas nowe cechy [3].
Bakteriofagi są to wirusy, które atakują i replikują się w komórkach bakteryjnych. Składają się one z materiału genetycznego otoczonego białkami strukturalnymi, które tworzą kapsyd. Materiałem genetycznym bakteriofagów jest DNA lub rzadziej RNA w postaci dwuniciowej lub jednoniciowej. Żaden z wirusów nie posiada obu rodzajów kwasu nukleinowego. Materiał genetyczny fagów zawiera od kilku do kilkuset genów kodujących białka niestrukturalne biorące udział w replikacji wirusa oraz strukturalne odpowiedzialne za budowę kapsydu. Genomy bakteriofagów znacznie różnią się między sobą wielkością. Materiał genetyczny fagów otoczony jest białkowym kapsydem, który jest odporny na niszczące działanie czynników zewnętrznych. Kapsyd jest złożony z podjednostek białkowych zwanych kapsomerami. Może on zawierać jeden rodzaj białka, ale zazwyczaj jest zbudowany z dwóch lub więcej rodzajów białek. Kapsyd może przybierać kształt heliakalny, izometryczny lub złożony, łączący w sobie obie te formy. 96% bakteriofagów posiada budowę złożona, a pozostałe 4% budowę helikalną i izomeryczną. Kapsyd bakteriofagów o złożonej strukturze jest zbudowany z wielościennej główki i ogonka. Głowa składa się z jednego lub kilku rodzajów białek i pełni funkcję ochronną kwasu nukleinowego, który znajduję się w jej wnętrzu. Od główki odchodzi ogonek, który u niektórych bakteriofagów ma zdolność kurczenia się, ułatwiającego wstrzykiwanie kwasu nukleinowego do wnętrza komórki bakteryjnej. Ogonek składa się z wewnętrznej tubularnej struktury rdzeniowej, otoczonej pochewką, przez którą następuje transfer kwasu nukleinowego podczas infekcji. Ogonek zakończony jest płytką podstawową, w którą wbudowane są białka enzymatyczne rozkładające ścianę komórkową bakterii. Od płytki podstawowej odchodzi sześć nitkowatych włókien białkowych, które służą do rozpoznawania odpowiednich receptorów oraz przytwierdzania się do powierzchni komórki bakteryjnej [5].
Bakteriofagi charakteryzują się wysoką specyficznością. Zazwyczaj jeden gatunek wirusa może namnażać się tylko w jednym gatunku bakterii lub w obrębie konkretnego jej szczepu. Namnażanie bakteriofagów może zachodzić w wyniku dwóch cyklów litycznego i lizygenicznego. Cykl lityczny polega na zakażeniu bakterii, namnożeniu nowych cząstek fagowych, rozpadzie komórki bakteryjnej i uwolnieniu nowych wirusów. Bakteriofagi wchodzące w cykl lityczny nazywane są wirulentnymi. Infekcja fagowa rozpoczyna się adsorpcją wirusa do powierzchni komórki bakteryjnej. W tym procesie ważną funkcję pełni płytka podstawowa oraz włókienka białkowe, które rozpoznają specyficzne receptory na powierzchni bakterii. Włókienka faga wiążą się w sposób odwracalny z receptorem bakteryjnym. Po przyłączeniu DNA bakteriofaga jest wstrzykiwane do cytoplazmy komórki bakteryjnej. W większości przypadków do komórki bakteryjnej wprowadzany jest tylko materiał genetyczny, natomiast pozostałe komponenty wirusa pozostają na zewnątrz. W zakażonej komórce bakteryjnej, fagowy materiał genetyczny może być replikowany przez polimerazę gospodarza, lub przez własną polimerazę, która jest syntetyzowana na podstawie informacji zawartej w genomie wirusowym. W wyniku tego procesu może powstać nawet kilkaset kopii fagowego DNA. Następnie na matrycy DNA powstałego w czasie replikacji, przy udziale rybosomów i enzymów bakterii, dochodzi do syntezy białek kapsydu. Powstałe białka spontanicznie łączą się z kopiami DNA i następuje składanie wirionów - kompletnych cząstek wirusowych. Komórka bakteryjna ulega lizie z uwolnieniem wielu cząsteczek fagowych, które mogą infekować inne komórki. Drugim rodzajem cyklu życiowego bakteriofagów jest cykl lizogeniczny, w którym genom faga integruje z genomem gospodarza. Cykl lizogeniczny polega na wnikaniu materiału genetycznego wirusa do komórki bakteryjnej i jego replikacji wraz z DNA gospodarza, nie powodując jego lizy. Bakteriofagi wchodzące w cykl lizogeniczny nazywane są łagodnymi lub umiarkowanymi. Cykl ten rozpoczyna się podobnie do cyklu litycznego, jednak różnica polega na tym, iż DNA faga po wniknięciu do komórki bakteryjnej ulega inaktywacji, a następnie włączeniu do genomu bakteryjnego w postaci tzw. profaga. Wirusowy DNA ulega replikacji wraz z DNA bakterii, dzięki czemu komórki potomne bakterii otrzymują również DNA wirusa. W cyklu lizogenicznym fag namnaża się w wyniku podziałów komórki bakteryjnej, nie powodując jej zniszczenia, natomiast bakterie zyskują dzięki obecności profaga oporność na dany rodzaj wirusa. W takiej formie bakteriofag może przetrwać nawet kilka podziałów. W określonych warunkach profag może ulec wycięciu z chromosomu bakteryjnego i wejść w cykl lityczny, który kończy się lizą komórki [5].
W zależności od typu bakteriofaga wyróżnia się transdukcję ogólną oraz specyficzną. Transdukcja ogólna zachodzi z udziałem fagów litycznych (wirulentnych), które po wprowadzeniu swojego materiału genetycznego degradują bakteryjne DNA do odcinków różnej długości (Rys.4). Po replikacji bakteriofaga, podczas składania fagów potomnych, do główki wirusa może zostać włączony, zamiast DNA fagowego, taki fragment bakteryjnego DNA, którego długość jest zbliżona do długości DNA wirusa. Powstaje wówczas bakteriofag zawierający w główce nie własny DNA, lecz przypadkowo pobrany fragment DNA bakteryjnego. Fag taki jest niezdolny do replikacji ale zachowuje zdolność adsorpcji do receptorów na powierzchni komórki bakteryjnej oraz wprowadzenia swojego materiału genetycznego. Zakażając inną bakterię, wprowadza do niej fragment DNA bakterii z której pochodzi. W ten sposób bakteriofag lityczny staje się wektorem przenoszącym DNA z jednej bakterii do drugiej. Włączenie fragmentu DNA do główki faga jest zdarzeniem przypadkowym, również fragment jest przypadkowy i może zawierać różne geny. Praktycznie każdy odcinek bakteryjnego DNA może zostać przeniesiony w ten sposób, stąd nazwa transdukcji ogólnej [3].
W większości przypadków DNA z komórki dawcy wprowadzone do cytoplazmy bakterii przez faga litycznego pozostaje w postaci pozachromosomalnej i przyjmuje strukturę kolistą, co zapobiega jego degradacji, ale również rekombinacji z chromosomem bakterii. Po każdorazowym podziale komórki, nie replikujący się DNA fagowy pozostaje w komórce macierzystej, a wszystkie powstające w wyniku podziałów komórki, nie posiadają go. Jeżeli taki fag transdukcje jakąś cechę, ujawnia się ona tylko w jednej komórce, która pierwotnie została zakażona. Wszystkie pozostałe komórki potomne nie posiadają tej cechy. Taki typ transdukcji nazywa się transdukcją poronną [3]. Około 8% nowo nabytych fragmentów DNA pozostaje w cytoplazmie bakterii w formie liniowej i ulega degradacji, a powstałe nukleotydy służą komórce bakteryjnej w procesach naprawy DNA. Jedynie około 2% nowo pozyskanych fragmentów DNA, homologicznych z genomem bakterii, ulega rekombinacji homologicznej z chromosomem [1].
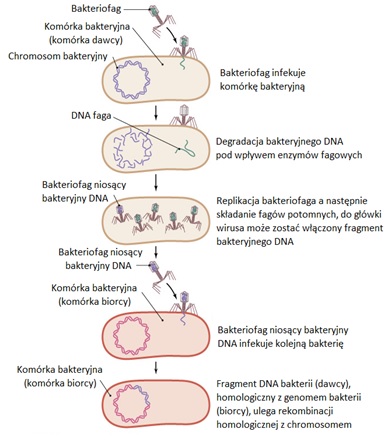
Rys.4. Proces transdukcji ogólnej u bakterii [6].
Transdukcja specyficzna dotyczy bakteriofagów łagodnych, których DNA po wprowadzeniu do komórki bakteryjnej ulega integracji z chromosomem bakterii i w tej postaci nazywane jest profagiem. Odłączający się z genomu bakterii, indukowany profag może przypadkowo wyciąć niewielkie fragmenty bakteryjnego DNA, które z nim sąsiadują. W wyniku, nowo powstające bakteriofagi będą zawierać poza fagowym DNA także fragment bakteryjnego DNA, z którego dany fag pochodzi. Zwykle wycięciu ulegają odcinki bakteryjnego DNA znajdujące się po jednej stronie wbudowanego genomu bakteriofaga, co oznacza, że aby zachować właściwą wielkość genomu, fag musi pozostawić jednakowy odcinek własnego DNA w genomie bakterii. Jeżeli pozostawiony w bakteryjnym genomie fragment wirusowego DNA nie posiada genów niezbędnych dla jego cyklu życiowego, materiał genetyczny bakteriofaga ulega replikacji z utworzeniem fagów potomnych zdolnych do zakażania kolejnych bakterii [1]. Wówczas może dojść do rekombinacji pomiędzy chromosomem zainfekowanej bakterii a DNA faga niosącym doczepione fragmenty DNA bakterii dawcy. W transdukcji tego typu bakteriofag przenosi geny sąsiadujące z miejscem integracji swojego DNA do chromosomu bakterii. Nazywamy ją transdukcją specyficzną, gdyż specyficznie przenoszone są tylko określone cechy [3]. Jeżeli jednak wyłączający się z genomu bakterii profag, przypadkowo wycinając fragment bakteryjnego DNA, pozostawi w genomie bakterii własne, niezbędne w procesie replikacji geny, utraci zdolność samodzielnego powielania się [1].
Obecność bakteriofagów w niemal każdym ekosystemie oraz ich zdolność przenoszenia materiału genetycznego pomiędzy bakteriami żyjącymi w różnych niszach ekologicznych przekłada się na ich ogromny wpływ na zmienność genetyczną bakterii [1]. Przenoszenie genów za pośrednictwem bakteriofagów, to najczęściej transfer odbywający się w obrębie jednego gatunku, aczkolwiek możliwa jest także międzygatunkowa wymiana informacji genetycznej. Horyzontalny transfer genów daje możliwość nabycia wielu genów w czasie jednego zdarzenia rekombinacyjnego. Jest to szczególnie istotne dla bakterii patogennych. Przykład mogą stanowić pałeczki Salmonella. Profagi obecne w genomie bakterii z rodzaju Salmonella modyfikują ich strukturę części O-antygenowej LPS, zmieniają faktory antygenów somatycznych, wpływają na wirulencję oraz warunkują znaczne zróżnicowanie pomiędzy serowarami Salmonella i wśród tego samego serowaru. Wyniki badań wskazują na stosunkowo dużą zawartość genów fagowych w genomach pałeczek Salmonella, co świadczy o istotnym udziale bakteriofagów w procesie ewolucji tych mikroorganizmów [7].
Transformacja
Proces transformacji został odkryty u pneumokoków przez Fredericka Griffitha w 1928 roku, a wyjaśniony w 1944 roku przez Oswalda Avery’ego. Polega on na pobieraniu przez bakterie materiału genetycznego ze środowiska. DNA pozyskiwane w ten sposób może wzbogacać informację genetyczną danego mikroorganizmu o nowe cechy [3].
Pobieranie zewnątrzkomórkowego DNA przez bakterie jest procesem złożonym (Rys.5). Bakteria może ulec transformacji tylko w określonych warunkach i okresach rozwojowych. Stan, w którym bakteria daje się transformować, nazywa się kompetencją. Kompetentne komórki bakteryjne posiadają na swojej powierzchni receptory wiążące fragmenty zewnątrzkomórkowego DNA oraz białka, chroniące pobrane DNA przed degradacją w cytoplazmie i umożliwiające rekombinacje pobranego DNA z genomem biorcy. Proces transformacji rozpoczyna się od związania pozakomórkowego fragmentu DNA z receptorem powierzchniowym kompetentnej komórki bakteryjnej. DNA obecne w środowisku najczęściej pochodzi z obumarłych komórek bakteryjnych, które rozpadając się uwalniają nieosłonięte cząsteczki kwasów nukleinowych. Ponadto, wiele bakterii może uwalniać swój materiał genetyczny do środowiska [1]. Kompetentne komórki mogą wiązać na swojej powierzchni dwuniciowe, stosunkowo niewielkie odcinki DNA o długości 7-9 kb, posiadające informację genetyczną równoważną paru genom [3]. Translokacja zewnątrzkomórkowego fragmentu DNA przez błonę komórkową jest odmienna u różnych gatunków bakterii. Najczęściej, po związaniu z receptorem powierzchniowym komórki biorcy, jedna z nici transformującego DNA ulega degradacji i do cytoplazmy biorcy wprowadzana jest pojedyncza nić DNA, dzięki czemu pobrany materiał genetyczny jest chroniony przed działaniem wewnątrzkomórkowych endonukleaz. Ponadto, ochronę pobranej nici DNA zapewniają białka znajdujące się w cytoplazmie komórki bakteryjnej. Pobrany fragment liniowej cząsteczki DNA jest włączany do genomu biorcy na zasadzie rekombinacji homologicznej pod warunkiem, że jest on komplementarny w przynajmniej 75% z DNA chromosomu biorcy [1].
Transformacja jest naturalnym procesem, podczas którego komórki bakteryjne zdobywają geny kodujące nowe cechy np. czynniki wirulencji, oporność na antybiotyki, na bakteriofagi, zdolność do fermentacji cukrów oraz syntezy aminokwasów i witamin [3]. Jeżeli kwas nukleinowy pobrany ze środowiska pochodzi od blisko spokrewnionego gatunku może być także wykorzystywany do naprawy uszkodzeń genomowego DNA lub stanowić źródło węgla, azotu i fosforu [1]. Najczęściej transformacja zachodzi w obrębie szczepów należących do tego samego gatunku, ze względu na dużą homologię odcinków DNA występującą pomiędzy spokrewnionymi osobnikami. Transformacja międzygatunkowa zachodzi rzadko i przede wszystkim miedzy gatunkami zbliżonymi do siebie. Wydajność procesu jest też wtedy znacznie mniejsza [3].
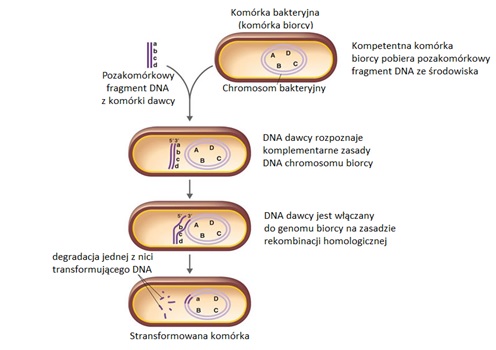
Rys.5. Proces transformacji u bakterii [8].
Słowa kluczowe: horyzontalny transfer genów, koniugacja, plazmid, transdukcja, bakteriofagi, cykl lityczny, cykl lizygeniczny, transformacja.
Autor: Katarzyna Czuba
Literatura:
1.Kasprzykowska U., Sobieszczańska B.M. Plastyczność bakteryjnych genomów – międzykomórkowy transfer informacji genetycznej. Post. Mikrobiol. 2014, 53, 2, 165–171.
2.Włodarczyk M. Co to jest plazmid? Kosmos. Problemy nauk biologicznych. 2002, 51, 3, 231-240.
3.Kunicki-Goldfinger W. J. H. Życie bakterii. Wyd. Nauk. PWN. 2006, 334-345.
4.http://biologyboom.com/conjugation-gene-recombination-in-e-coli/ (zmodyfikowano).
5.Winter P. C., Hickey G. I., Fletcher H. L. Krótkie wykłady-Genetyka. Wyd. Nauk. PWN. 2000.
6.http://www.quazoo.com/q/Transduction_%28genetics%29 (zmodyfikowano).
7.Dera-Tomaszewska B. Plazmidy i bakteriofagi występujące u bakterii Salmonella. Post. Mikrobiol. 2011, 50, 3, 201–208.
8. http://classes.midlandstech.edu/carterp/Courses/bio225/chap08/lecture6.htm (zmodyfikowano).
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje