

|
Zamknij X
|
Wstęp
Grypa jest ostrą chorobą zakaźną układu oddechowego wywołaną zakażeniem wirusem grypy. Wirusy grypy należą do rodziny Ortomyksowirusów. Znane są trzy typy wirusów grypy A, B oraz C, z których wi-rus typu A jest najlepiej poznany. Wirus grypy typu A dzieli się na podtypy ze względu na występujące w otoczce glikoproteiny powierzchniowe, neuraminidazę (NA) i hemaglutyninę (HA). Jak dotąd poznano 9 odmian NA i 16 odmian HA, co daje łącznie 144 możliwych kombinacji segmentów genowych i powoduje istnienie ogromnej różnorodności wirusów typu A. Wirus grypy A występuje zarówno u ludzi jak i zwierząt. Naturalnymi gospodarzami wirusa grypy typu A są dzikie ptaki wodne oraz lądowe. Wykryto u nich ponad 100 różnych kombinacji glikoprotein powierzchniowych. Tylko 3 z tych podtypów (H1N1, H2N2, H3N2) przenoszą się od człowieka do człowieka [1].
Wirus grypy typu A jest wirusem podwyższonego ryzyka, corocznie wywołuje epidemie i sporadycznie pandemie. Epidemie grypy każdego roku powodują śmierć około 250-500 tysięcy ludzi na całym świecie. Jedną z najbardziej znanych pandemii wywołanych wirusem grypy typu A była tzw. grypa hiszpanka w latach 1918-1919. Spowodowała ona śmierć około 50 milionów ludzi. Szczepy wywołujące pandemie charakteryzują się wysoką śmiertelnością oraz mają zdolność do szybkiego rozprzestrzeniania się [2]. Obecnie istotne zagrożenie stanowią dwa szczepy wirusa: H5N1 (wirus ptasiej grypy) oraz H1N1 (wirus świńskiej grypy). Przenoszenie się wirusa H5N1 z drobiu na człowieka zaobserwowano w 15 krajach Azji i Afryki. Natomiast nie odnotowano przenoszenia się tego szczepu wirusa bezpośrednio między ludźmi [3].
Wirus grypy podlega dwóm podstawowym mechanizmom ewolucji. Pierwszy z nich, przesunięcie genetyczne, jest przyczyną epidemii, powodując konieczność produkcji nowych szczepionek przeciw aktualnie występującym szczepom wirusa. Przesunięcie genetyczne związane jest ze stopniową akumulacją punktowych mutacji w genomie pojedynczego szczepu, które powodują zmiany w determinantach antygenowych. Wówczas wirus zyskuje częściową lub całkowitą odporność na odpowiedź układu immunologicznego gospodarza. Drugi mechanizm, skok antygenowy, skutkuje pojawieniem się nowego szczepu wirusa, który może wywołać pandemię. Skok antygenowy następuje w wyniku przenoszenia się wirusa pomiędzy różnymi gatunkami. W międzygatunkowym przemieszczaniu się wirusa grypy typu A biorą udział świnie, które są podatne na iniekcję zarówno wirusem pochodzenia ludzkiego, jak i ptasiego. Podczas zakażenia dwoma szczepami wirusa może nastąpić reasortacja 8 niezależnych segmentów RNA, z których złożony jest genom wirusa grypy typu A. Na skutek wymieszania się segmentów pochodzących od różnych szczepów istnieje możliwość pojawienia sie 256 różnych genotypów [2].
Budowa wirusa grypy
Wiriony grypy najczęściej mają kształt sferyczny lub tworzą formy włókniste. Średnica pojedynczego wirionu wynosi ok. 100 nm. Otoczkę wirusa stanowi dwuwarstwa lipidowa, w którą wbudowane są białka transbłonowe: neuraminidaza (NA), hemaglutynina (HA) oraz kanał jonowy M2. Hemaglutynina stanowi 80% białek otoczki, nauraminidaza około 17%, natomiast kanał jonowy M2 3% białek otoczki. Białka transbłonowe tworzą na powierzchni wirionu charakterystyczne wypustki (Rys.1). Otoczka lipidowa, pochodząca z błony komórkowej gospodarza, zawiera rafty lipidowe bogate w choresterol oraz regiony płynnej błony „nieraftowej". Z domeną raft lipidowych związane są hemaglutynina i neuraminidaza, natomiast z „nieraftową" częścią błony - kanał jonowy M2. Po wewnętrznej stronie otoczki znajduje się białko błonowe M1, które zapewnia właściwą strukturę wirionu oraz oddziałuje z rdzeniem wirusa. Rdzeń wirusa zbudowany jest z ośmiu kompleksów rybonukleoproteinowych (vRNP). Pojedynczy vRNP składa się z nici RNA o ujemnej polarności (vRNA), nukleoproteiny (NP), polimerazy (PA1, PB2, PA) oraz niewielkich ilości białka NEP. Służy on jako matryca dla wirusowej polimerazy. Cząsteczki RNP stanowią niezależne jednostki replikacyjno-transkrypcyjne, w obrębie, których polimeraza prowadzi oba procesy. Genom wirusa jest złożony z ośmiu cząsteczek RNA kodujących 11 białek [4].
Nukleoproteina związana z vRNA ma istotny wpływ na strukturę drugorzędową materiału genetycznego wirusa. Wiąże ona vRNA, co może powodować rozplecenie nici. Struktury drugorzędowe vRNA wirusa grypy mają funkcjonalne znaczenie podczas cyklu replikacyjnego wirusa. W zależności od etapu cyklu rozwojowego wirusa, częściowo komplementarne końce 5' i 3' mogą tworzyć dynamiczne struktury drugorzędowe. W wyniku tego procesu powstaje struktura pętli z dwoma odseparowanymi wybrzuszeniami, tzw. struktura panhandle, która przypomina podwójną helisę typu A. Struktura ta determinuje zamkniętą konformację vRNA. Wykazano jednak, że struktura panhandle nie jest wymagana do inicjacji transkrypcji, co doprowadziło do powstania modelu typu RNA-fork. Model ten jest zmodyfikowaną strukturą panhandle, który tylko częściowo jest dwuniciowy. Kolejny model, corkscrew, posiada dwie małe „spinki" na końcach vRNA. Struktura ta jest wymagana do przylegania kompleksu polimerazy i uzyskania przez ten kom-pleks aktywności endonukleazowej. Oprócz tego „spinka" przy końcu 5' vRNA w częściowo rozplecionej strukturze corkscrew, czyli modelu hook, jest niezbędna w procesie poliadenylacji mRNA. Poliadenylacja polega na powtarzającym się kopiowaniu ciągu 5-7 reszt urydyny, które sąsiadują z rdzeniem panhandle. „Spinka" w strukturze hook, może tworzyć barierę, która powoduje zatrzymanie polimerazy, podczas syntezy mRNA. Na skutek tego powstaje ogon poli(A) [5].
Struktura fragmentów końcowych vRNA ma istotne znaczenie w procesie replikacji i transkrypcji. W różnych etapach cyklu wirusa vRNA struktura panhandle ulega konformacyjnym przemianom pomiędzy strukturami drugorzędowymi wymaganymi do różnych funkcji. Przełączenie między otwartą a zamkniętą formą vRNP jest regulowane poprzez nukleoproteiny i polimerazy. Białko NP rozplata strukturę RNA, podczas gdy polimeraza ją usztywnia [5].
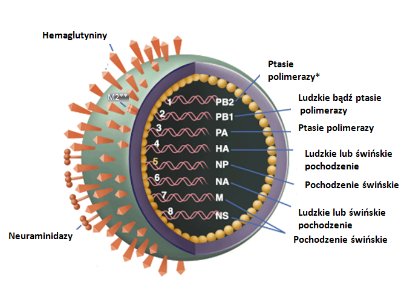
Rys.1. Budowa wirusa grypy [24].
Białko PB1 stanowi jedną z podjednostek heterotrimeru tworzącego wirusową polimerazę RNA. Znajduje się w centrum kompleksu i oddziałuje z każdą z pozostałych podjednostek, PB2 i PA. Białko PB1 zawiera rdzeń katalityczny RNA polimerazy RNA – zależnej (RdEp). Odpowiada ono za syntezę wirusowego mRNA oraz replikację całego genomu wirusa [6].
Białko PB1-F2 zawiera 87 reszt aminokwasowych oraz posiada amfipatyczną helisę zlokalizowaną przy jego końcu C. Helisa ta kieruje białko do mitochondriom. PB1-F2 powoduje zmiany w przepuszczalności błony mitochondrialnej, prowadząc do śmierci komórki. Zjawisko apoptozy występuje głównie w komórkach układu immunologicznego. Białko PB1-F2 wywołuje apoptozę monocytów, wzmaga stan zapalny i podnosi ryzyko wtórnego zapalenia płuc [6].
Białko PB2 stanowi składnik wirusowej polimerazy RNA. W jądrze komórkowym PB2 rozpo-znaje i wiąże fragmenty kap przy końcach 5' mRNA komórki gospodarza. Struktury kap, po odcięciu przez domenę PA, mogą służyć jako startery do transkrypcji wirusowego mRNA. Podjednostką PB2 może stanowić determinantę wirulencji podtypu H5N1 [7].
Białko PA stanowi podjednostkę wchodzącą w skład kompleksu wirusowej polimerazy. Wykazuje aktywność proteazową i endonukleazową. Domena endonukleazową na N końcu, w miejscu aktywnym, zawiera aminokwasy, które biorą udział w katalitycznej reakcji odłączenia struktur kap od mRNA gospodarza. Uzyskane w ten sposób fragmenty kap służą jako statery w procesie transkrypcji wirusowego mRNA (cap-snatching) [7].
Hemaglutynina (HA) jest powierzchniowym białkiem wirusa. Odpowiada ona za rozpoznawanie receptorów glikoproteinowych lub glikolipidowych obecnych na powierzchni komórek nabłonkowych układu oddechowego gospodarza. Głównym składnikiem odpowiedzialnym za rozpoznanie receptorów przez hemaglutyninę jest kwas sjalowy (SA). Hemaglutynina bierze także udział w fuzji otoczki lipidowej wirionu z błoną komórkową. Hemaglutynina jest związana z otoczką lipidową wirusa tworząc homotrimer. Proteazy układu oddechowego gospodarza są zdolne do rozcięcia HA na podjednostki HA1 i HA2, prowadząc do uzyskania infekcyjności przez wirusa. Podjednostka HA1 stanowi miejsce wiązania kwasu sialowego. W tej części białka znajdują się determinanty antygenowe (epitopy), przeciw którym układ odpornościowy gospodarza wytwarza immunoglobuliny podczas infekcji. Natomiast podjednostka HA2 tworzy część transbłonową kotwiczącą białko w otoczce lipidowej wirusa i uczestniczy w fuzji pomiędzy wirusową osłonką i błoną endosomu. Miejsce cięcia hemaglutyniny jest zależne od sekwencji aminokwasów i zachodzi jedynie przez zewnątrzkomórkowe trypsynopodobne proteazy, znajdujące się w górnych partiach dróg oddechowych lub w układzie pokarmowym. W ten sposób dochodzi jedynie do infekcji lokalnej. Hemaglutynina wysoce wirulentnych podtypów H5 i H7 zawiera kilka reszt aminokwasowych w miejscu cięcia cząsteczki, które są rozpoznawane przez liczne proteazy gospodarza, co prowadzi do infekcji ogólnoustrojowej [8].
Neuraminidaza (NA) stanowi białko powierzchniowe wirusa grypy. Katalizuje ona reakcję odcięcia reszt kwasu sialowego z glikoprotein i glikolipidów zewnętrznej części błony komórkowej gospodarza oraz z nowo powstałych cząsteczek wirusowych. Zaburzenie tego procesu skutkuje wiązaniem się hemaglutyniny z kwasem sialowym, co prowadzi do powstania agregatów komórkowo-wirusowych. Wówczas zostaje utrudnione uwalnianie potomnych wirionów z powierzchni błony [6].
Nukleoproteina (NP) jest białkiem strukturalnym, związanym z pojedynczym segmentem RNA oraz polimerazą tworząc rybonukleoproteinę (RNP). Nukleoproteina uczestniczy w regulacji aktywności replikacyjno-transkrypcyjnej wirusowej polimerazy RNA. Może ona wiązać się z podjednostkami polimerazy PB1 i PB2 powodując rozpoczęcie replikacji, nie wymagającej starterów. Oprócz tego nukleoproteina uczestniczy w oddziaływaniu z komórkowymi polipeptydami, jak aktyna czy białka odpowiedzialne za import i eksport jądrowy [9].
Białko M1 jest głównym białkiem strukturalnym determinującym morfologię wirionu. Tworzy ono sieć helis białkowych z systemem wolnych przestrzeni, które są miejscem oddziaływań z cytoplazmatycznymi domenami NA i HA. Białko M1 opóźnia proces pączkowania, indukowany przez hemaglutyninę, do momentu upakowania cząsteczek niezbędnych do funkcjonowania wirusa. M1 bierze udział w pakowaniu cząsteczek wirusowych, pączkowaniu i uwalnianiu potomnych wirionów. Oddziałuje ono z warstwą lipidową, powodując asymetrię błony komórkowej gospodarza wymaganą w procesie inicjacji pączkowania. Białko M1 może także wiązać NP, które jest zawarte w kompleksie rybonukleoproteinowym. Zmniejszanie wbudowywania M1 może zakłócać rekrutację RNP i zmieniać strukturę wirionu [10].
Białko M2 (białko kanału jonowego) jest integralnym białkiem błonowym, tworzącym tetramery w otoczce lipidowej wirusa. Posiada ono domenę trans-membranową (TM) i cytoplazmatyczną amfipatyczną helisę. Zachowawcze aminokwasy His37 i Trp41, znajdujące się w domenie budującej kanał jonowy, są istotnymi elementami dla transportu protonów. Trp41 w pH zbliżonym do neutralnego tworzy bramę zamykającą kanał. Kanał otwiera się w niskim pH w związku ze zmianami informacyjnymi TM. Obniżenie pH skutkuje protonacją His37, co zwiększa przepływ protonów przez kanał jonowy. Dochodzi wówczas do zakwaszenia wnętrza wirionu. Pozwala to na uwolnienie zawartości wirionu do cytoplazmy i kontrolę pH w aparacie Golgiego pod-czas syntezy hemaglutyniny [6].
Białko NS1 tworzy homodimer, który nie wchodzi w skład wirionu. Domena N-terminalna bierze udział w wiązaniu dwuniciowego RNA (dsRNA). Wychwytywanie dsRNA zapobiega indukcji syntetazy 2'-5'-oligoadenylowej. Na skutek tego zostaje zahamowana aktywność wewnątrzkomórkowej RNazy L, która hydrolizuje komórkowe i wirusowe RNA. Prowadzi to do apoptozy komórki przed zakończeniem cyklu replikacyjnego wirusa. Białko NS1 stanowi ważną determinantę wirulencji. Ma ono również związek z wysokim poziomem cytokin prozapalnych i obniżonym poziomem cytokin antyzapalnych. Deregulacja odpowiedzi układu odpornościowego wpływa na ostry przebieg choroby [11].
Białko NS2, nazywane również NEP, ponieważ jest odpowiedzialne za eksport jądrowy rybonukleoprotein oraz M1. N-terminalny fragment białka NS2 oddziałuje z CRM1 - eksportyną związaną z transportem jądrowym. Eksportyną CRM1 bierze udział w przeniesieniu całego kompleksu z jądra do cytoplazmy komórki. Natomiast C-terminalny fragment NS2 wiąże białko M1 zaangażowane w oddziaływanie z nukleoproteiną zawartą w kompleksie vRNP. Białko NS2 uczestniczy również w regulacji transkrypcji i replikacji wirusowej. NS2 generuje powstawanie krótkich niekodujących RNA (svRNA), które mogą brać udział w regulacji przełączania aktywności transkrypcyjnej na replikacjną [12].
Cykl życiowy wirusa grypy
Wirus grypy typu A atakuje komórki nabłonka górnych dróg oddechowych, w których się namnaża (Rys.2). W pierwszym etapie zakażenia hemaglutynina znajdująca się na powierzchni otoczki wirusowej rozpoznaje i wiąże receptory glikoproteinowe lub glikolipidowe obecne na powierzchni komórek gospodarza. Głównym składnikiem odpowiedzialnym za rozpoznanie receptorów przez hemaglutyninę jest kwas sjalowy (SA2,3aGal oraz SA2,6aGal). Następnie wirus wnika do wnętrza komórki gospodarza w drodze endocytozy na skutek wpuklenia błony komórkowej i odcięcia pęcherzyka endocytarnego (endosomu). Wyniki badań sugerują, że za mechanizm w wyniku, którego wirus wyzwala endocytozę, są odpowiedzialne sferyczne cząsteczki wirusowe. Wiążą się one z powierzchnią komórki, powodując aktywację receptora o aktywności kinaz tyrozynowych. Aktywacja receptora wywołuje kaskadę sygnałów prowadzącą do formowania de novo jamek klatrynowych (clathrin pits), które stanowią miejsca endocytozy zależnej od klatryny [13].
Po wniknięciu wirusa do komórki, niskie pH w endosomie powoduje otwarcie kanału jonowego M2 i przepływ protonów w poprzek otoczki wirusowej. Obniżenie pH we wnętrzu wirionu skutkuje oddzieleniem vRNP od białka M1. Na powierzchni białka NP następuje odsłonięcie sygnału lokalizacji jądrowej (NLS). Warunki panujące w endosomie mają także wpływ na zmiany konformacyjne w rozciętej cząsteczce hemaglutyniny (HA). Dochodzi do wbudowania w membranę endosomu N-terminalnej domeny HA2. W wyniku tych przemian dochodzi do fuzji otoczki wiru-sowej z błoną endosomalną. Kompleksy rybonukleoproteinowe zostają uwolnione do cytoplazmy. Wszystkie podjednostki wirusowej polimerazy RNA (PB1, PB2, PA) oraz białko NP posiadają sygnał lokalizacji jądrowej. Transport do jądra komórki gospodarza odbywa się przez kompleksy porów jądrowych (NPC). Komórkowe importyny α i β są zaangażowane w transport jądrowy. Rozpoznają one NLS znajdujące się na cząsteczkach RNP. Wówczas zostaje utworzony kompleks RNP/importynaα/importynaβ, który na drodze transportu aktywnego zostaje przeniesiony do jądra poprzez NPC [13,14].
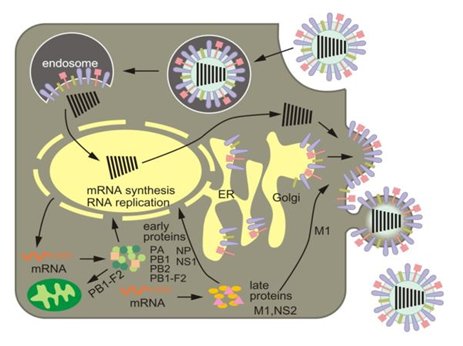
Rys. 2. Schemat cyklu życiowego wirusa grypy typu A [2].
W komórkach zainfekowanych wirusem grypy typu A zostaje zatrzymana synteza białek gospodarza, a następuje translacja wirusowego mRNA. W jądrze komórkowym wirusowa polimeraza RNA rozpoczyna syntezę wirusowego mRNA. Istotnym etapem tego procesu jest cap-snatching, w którym wirusowa polimeraza RNA zależna od RNA (RdRp) wiąże się z fosforylowaną komórkową polimerazą II. Ufosforylowana polimeraza II aktywuje syntezę czapeczki guanozynowej (kap) na 5' końcu mRNA gospodarza [13,14]. Podjednostka białka PB2 wiąże strukturę kap mRNA, a domena o aktywności endonukleazowej w podjednostce białka PA odcina 10-13 reszt nukleotydowych za czapeczką guanozynową. Odcięty od mRNA komórkowego fragment kap służy jako starter do syntezy wirusowego mRNA [6,13]. W procesie dojrzewania wirusowego mRNA, NS1 przyłącza się do U6 i innych komponentów splicingowych, powodując ich przeniesienie do jądra zainfekowanej komórki. Istotną rolę w procesie redagowania mRNA wirusa ogrywa również białko NP. Oddziałuje ono z czynnikiem dojrzewania UAP56, który ma aktywność ATPazy oraz aktywność helikazy RNA zleżnej od ATP. Białko NP zaangażowane jest również w eksport mRNA z jądra [13,14]. Poliadenylacja wirusowego mRNA następuje przez kopiowanie ciągu urydyn znajdujących się na 5' końcowych rejonach każdego segmentu. Dojrzałe wirusowe mRNA są przenoszone do cytoplazmy i tam ulegają translacji z udziałem aparatu translacyjnego komórki. Pierwszym powstają białka NP, NS1, PB1, PB2 i PA, które migrują do jądra komórkowego. Następnie powstają białka powierzchniowe HA, NA i M2, które ulegają modyfikacjom w retikulum endoplazmatycznym. Po glikozylacji, która zachodzi w aparacie Golgiego, wszystkie białka kierowane są do błony komórkowej [6,13].
Kolejnym etapem cyklu życiowego wirusa jest powielanie materiału genetycznego. Istnieje wiele hipotez wyjaśniających proces przełączania aktywności polimerazy z transkrypcyjnej na replikacyjną. Najbardziej prawdopodobny model regulacji cis-łrans zakłada, że polimeraza obecna w vRNP, działając in cis, jest odpowiedzialna za transkrypcję. Natomiast po wniknięciu do jądra, nowo powstałe podjednostki polimerazy, rozpoczynają replikację vRNA (działanie in trans) [13,14]. Powielanie genomu następuje w dwóch etapach. W pierwszym syntetyzowana jest nić cRNA, która jest komplementarna do vRNA. Następnie replikon o dodatniej polarności (cRNP) stanowi matrycę do syntezy potomnych vRNA. Powstałe vRNA wraz z nowo zsyntetyzowanymi białkami tworzy kompleksy vRNP, które są eksportowane do cytoplazmy. W transporcie bierze udział białko M1, bezpośrednio oddziałujące z vRNP, oraz białko NEP (NS2), które maskuje sygnał NLS w M1. Białko NEP (NS2) zawiera sekwencję eksportu jądrowego (NES), która jest ważnym elementem transportu przez błonę jądrową. Rozpoznaje ono białko CRM1, które wiąże substrat (NEP-M1-vRNP) w obecności Ran-GTP. Wówczas powstaje potrójny kompleks, który po hydrolizie GTP, kontrolowanej przez RanGAP i RanBP1, jest uwalniany do cytoplazmy [13,15].
W pakowaniu segmentów do wirionu ważną funkcję pełni struktura fragmentów końcowych vRNA. Jedna kopia każdego segmentu jest pakowana do pojedynczej cząsteczki wirusa. Istnieją dwa modele mechanizmu składania. Pierwszy model przypadkowego pakowania, sugeruje, że wszystkie segmenty posiadają wspólny sygnał kierujący je do wnętrzna nowo powstającego wirionu. Drugi model, selektywnego pakowania, zakłada, że wszystkie osiem segmentów vRNA posiada unikatowy sygnał do wbudowywania w winion. Zróżnicowane w obrębie segmentów sygnały pakowania mogą służyć do powstawania wielosegmentowej cząsteczki, która ułatwia wbudowywanie zbioru ośmiu vRNA do wirionu [5].
Nukleoproteina (NP) oddziałuje z mikrofilamentami aktynowymi, które są odpowiedzialne za transport do apikalnego bieguna komórki. Mikrofilamenty aktynowe transportują kompleksy M1-vRNP do miejsca uwalniania nowo powstałych cząstek wirusowych. Powodują wzrost wirionu przez „wpychanie" do jego wnętrza vRNP. W wyniku czego powstaje siła „wypychająca" pączek, natomiast siłę „wyciągającą" stanowią białka zmieniające krzywiznę błony. Istotną rolę w wbu-dowywaniu vRNP do wirionów potomnych odgrywają białka M1 i NP. Podczas pączkowania M1 oddziałuje z M2, powodując umiejscowienie białka M2 w obrębie tratwy lipidowej. Umożliwia to kompletne upakowanie vRNA wraz z białkami oraz wpływa na stabilizację procesu pączkowania. Istotnym elementem w odcinaniu i uwalnianiu wirionów potomnych jest białko M2. Znajduje się ono na granicy dwóch faz. W tratwie lipidowej, na szyjce pączkującego wirionu, prowadzi do wpuklenia, natomiast w rejonie bardziej płynnej części błony powoduje uwypuklanie membrany. Na skutek modyfikacji napięcia między fazami dochodzi do odcięcia i uwolnienia cząstek wirusa [13,14].
Po ukształtowaniu się wirionu neuraminidaza (NA) katalizuje reakcje odcięcia kwasu sialowego z powierzchni glikoprotein znajdujących się na błonie komórkowej i na otoczce wirusa. W konsekwencji potomne wiriony zostają uwolnione z powierzchni komórki [13,16].
Podczas cyklu życiowego wirusa mogą powstawać zarówno cząsteczki infekcyjne, jak i nieinfekcyjne. Ponad 90% wirusów potomnych nie jest zakaźna. Uszkodzenia zaburzające funkcjonalność wirionów powstają na skutek nieprawidłowej budowy białek wirusowych oraz z wadliwego pakowania wirionów [10,13].
Perspektywy terapii wirusa grypy typu A
Zmienność wirusa grypy typu A stanowi przyczynę corocznych epidemii oraz pojawiających się rzadziej pandemii. Niepokojące jest także pojawianie się nowych szczepów wirusa, wykazujących zdolność przenoszenia na kolejnych gospodarzy. Prowadzonych jest wiele badań nad budową i funkcją wirusowych białek oraz genomu. Poznanie mechanizmów odpowiedzialnych za namnażanie wirusa umożliwiłoby projektowanie leków przeciwwirusowych o szerokim spektrum działania. Natomiast terapeutyki ukierunkowane na inhibicję zachowawczych ewolucyjne mechanizmów regulacji mogłyby zwalczać liczne podtypy wirusa grypy. Jednocześnie zastosowanie takich leków przeciwwirusowych umożliwiłoby zmniejszenie ryzyka powstawania opornych mutantów [6,13].
Istotnym etapem w tworzeniu wirusowych mRNA jest odcinanie fragmentu kap od mRNA gospodarza. Uzyskane w ten sposób fragmenty kap służą, jako statery w procesie transkrypcji wirusowego mRNA. Ważną rolę w tym procesie odgrywa domena endonukleazowa białka PA, która bierze udział w katalitycznej reakcji odłączenia struktur kap od mRNA gospodarza. Reakcja ta przebiega w obecności jonów metalu. Kwas 2,4-diokso-4-fenylobutanowy, wiąże jony metalu biorące udział w procesie katalizy. Wówczas pierścień fenylowy zostaje umiejscowiony w rowku rdzenia katalitycznego tworząc blokadę steryczną. Zamiana pierścienia fenylu na większą grupą funkcyjną wzmacnia właściwości inhibicyjne. Kwas 2,4-diokso-4-fenylobutanowy wykazuje specyficzność względem podjednostki PA, nie wywierając hamującego wpływu na polimerazy komórkowe ani na aktywność innych nukleaz, co czyni go potencjalnym terapeutykiem [13,17].
Integralność kompleksu polimerazy gwarantuje wydajne jego funkcjonowanie. Prowadzone są badania nad oddziaływaniami pomiędzy podjednostkami PA, PB1 i PB2 w kompleksie polimerazy. Wykazano, że ostatnie 14 reszt aminokwasowych domeny N-terminalnej PB1 uczestniczy w wiązaniu rejonu C-terminalnego PA. Aminokwasy odpowiedzialne za te od-działywania są wysoce zachowawcze w wirusach H5N1 (wirus ptasiej grypy) oraz H1N1 (wirus świńskiej grypy). Rozwiązanie struktury rejonu C-terminalnego PB1 i rejonu N-terminalnego PB2 ujawniło, że mutacje w obszarze wzajemnego oddziaływania tych podjednostek hamują syntezę RNA. Związki, które uczestniczą w dysocjacji podjednostek PB1-PB2, stanowią potencjalne inhibitory dla wirusa grypy [13,18].
Hemaglutynina (HA) jest związana z otoczką lipidową wirusa. Odpowiada ona za rozpoznawanie receptorów glikoproteinowych lub glikolipidowych znajdujących się na powierzchni nabłonka układu oddechowego gospodarza, a także doprowadza do fuzji z błoną endosomu. Tert--butylohydrochinon (TBHQ) wiążąc się z hemaglutyniną, zapobiega jej zmianie konformacyjnej i wbudowaniu domeny HA2 w błonę endosomu. Zapobiega to uwolnieniu RNA wirusa do cytoplazmy komórki. Inne modyfikacje grupy tert-butylowej mogłyby poprawić hydrofobowe oddziaływania w obrębie kieszeni wiążącej, a dodatkowe polarne oddziaływania przyczyniłyby się do podniesienia skuteczności inhibitora [13,19]. Podobne działanie wykazują ludzkie przeciwciała monoklonalne CR6261. Immunoglobiny rozpoznają i wiążą błonowy region HA1 oraz HA2. W ten sposób zapobiegają konformacyjnym przemianom prowadzącym do fuzji otoczki wirusa z błoną endosomu. Ludzkie przeciwciała monoklonalne CR6261 mogłyby stać się ważnym elementem terapii przeciwgrypowej [20].
Neuraminidaza katalizuje reakcję odcięcia reszt kwasu sialowego z glikoprotein i glikolipidów nowo powstałych cząsteczek wirusowych. Zaburzenie tego procesu powoduje wiązanie się hemaglutyniny z kwasem sialowym. Wówczas zostaje utrudnione uwalnianie potomnych wirionów z powierzchni błony. Zanamivir i oseltamivir są inhibitorami neuraminidazy (Rys.3). Zanamivir jest analogiem kwasu sialowego, w którym na miejsce grupy hydroksylowej podstawiona jest grupa guanidynowa. W oseltamivirze część glicerolowa podstawiona jest hydrofobową grupą L-etylo-propoksylową, a część guanidynowa - grupą aminową. Niektóre szczepy wirusa grypy nabyły oporność na oseltamivir. Neuraminidaza zawierająca związany oseltamivir wykazuje zmianę strukturalną w pobliżu miejsca wiążącego kwas sialowy. Oznacza to, że neuraminidaza ulega zmianom konformacyjnym po przyłączeniu substratu. Daje to możliwość projektowania nowych podstawników kwasu sialowego lub allosterycznych inhibitorów wiążących się z przejściową strukturą NA [6,13].
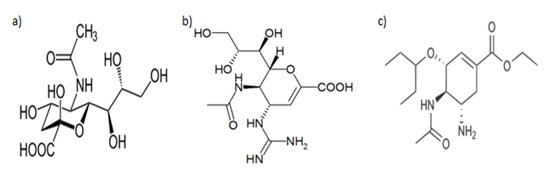
Rys.3. Wzory strukturalne (a) kwasu sialowego i jego analogów stosowanych jako inhibitory neuraminidazy: zanamivir (b) i oseltamivir (c) [6].
Nukleoproteina jest niezbędna podczas procesu replikacji. Uczestniczy ona w regulacji aktywności replikacyjno-transkrypcyjnej wirusowej polimerazy RNA. Wykazano, że nukleozyna wiążąc się z jedną z domen nukleoproteiny, wpływa na zmianę konformacji białka. Zmiany te indukują formowanie dużych agregatów nukleoproteiny w cytoplazmie komórki. W wyniku czego dochodzi do całkowitego zahamowania transportu nukleoproteiny do jądra komórkowego [6,13].
Białko M2 stanowi kanał jonowy, który pełni istotne funkcje w licznych etapach cyklu życiowego wirusa. Hamujące działanie względem funkcji białka M2 wykazują amantadyna i rymantadyna. Amantadyna jest umiejscowiona w otoczeniu reszt aminokwasowych, stanowiąc steryczną blokadę kanału. Natomiast rymantadyna blokuje kanał jonowy poprzez allosteryczną inhibicję. Związek ten wchodzi w oddziaływania z resztami aminokwasowymi, w konsekwencji stabilizując i utrzymując transmembranową domenę białka w stanie zamkniętym. Amantadyna podobnie jak rymantadyna, może wywoływać szybkie wytwarzanie oporności wirusów [6,13].
Białko NS1 wpływa na podwyższony poziom cytokin prozapalnych i obniżony poziom cytokin antyzapalnych. NS1 blokuje czynnik specyficzności cięcia i poliadenylacji (CPSF). Białko NS1 wiąże dwa palce cynkowe: F2F3. Mechanizmy te hamują odpowiedź odpornościową komórki. Małe cząsteczki zawierające na trzy aromatyczne reszty aminokwasowe, przypominające strukturę F3, mogłyby wychwytywać białka NS1 i skutecznie hamować wiązanie NS1 z CPSF [6,13].
Prowadzone są również badania nad zaprojektowaniem cząsteczek, które hamowałyby wirusową amplifikację, poprzez oddziaływanie z RNA. Próby zastosowania antysensowych oligonukleotydów nakierowanych na RNA wirusa grypy stwarzają możliwość selektywnego wyłączania genów oraz eliminację ich produktów. W tym celu wykorzystano rybozymy, cząsteczki rozpoznające odpowiednią sekwencję RNA, a następnie rozcinające ją w drodze reakcji enzy-matycznej. Niestety rybozymy charakteryzują się niską stabilnością. Właściwości te mocno ograniczałyby ich terapeutyczne zastosowanie [13,21].
Obiecujące możliwości projektowania leków przeciwwirusowych stwarzają DNazymy. Charakteryzują się one wyższą stabilnością i wydajnością niż antysensowe oligonukleotydy. DNazymy zawierają sekwencję komplementarną do regionu mRNA PB2, kodującego miejsce odpowiedzialne za rozpoczęcie translacji. Aktywność przeciwwirusowa takich katalitycznych cząsteczek jest bardzo wysoka i nakierowana na inhibicję namnażania wirusa grypy [13,21].
Zadowalające wyniki dostarczają także badania prowadzone na antysensownych oligomerach. Oddziaływują one w sposób sekwencyjnie specyficzny, redukując miano wirusa o 85%. Fosforodiamidowe oligomery morfolinowe (PMO) są strukturalnie podobne do jednoniciowego DNA. Jednostką podstawową łańcucha jest zasada purynowa lub pirymidynowa połączona z pierścieniem morfolinowym, który jest związany z fosforodiamidem. PMO są odporne na działanie nukleaz. Oligomery te zakłócają ekspresję genów poprzez tworzenie dupleksów z komplementarnym RNA. W ten sposób powstaje steryczna blokada. Wniknięcie do komórki jest możliwe dzięki związaniu oligomeru z peptydem bogatym w argininy (ARP). Powstały koniugat peptyd-PMO (P-PMO) jest wykorzystywany w leczeniu infekcji wywołanych wirusem grypy. Aktywność przeciwwirusowa została zaobserwowana dla różnych podtypów wirusa grypy: H1N1, H3N1, H3N8, H5N1, H7N7 [13,22].
W projektowaniu leków przeciw wirusowi grypy typu A wykorzystano także zjawisko interferencji RNA. Interferencja RNA (RNAi) jest naturalnym procesem, którego jedną z funkcji jest ochrona genomu przed wirusami i transpozonami. Proces RNAi polega na posttranskrypcyjnym wyciszaniu genów z zastosowaniem krótkich dwuniciowych cząsteczek RNA (dsRNA). W pierwszym etapie RNAi dochodzi do enzymatycznej hydrolizy dsRNA, przez enzym Dicer. W wyniku tego procesu powstają krótkie, dwuniciowe siRNA. Następnie powstałe siRNA są przyłączane do zależnego od RNA kompleksu wyciszającego RISC, który degraduje sensowną nić RNA w cząsteczce siRNA. Antysensowa nić siRNA wraz z kompleksem białkowym RISC przyłącza się do docelowego mRNA i powoduje jego degradację. Podjęto próby zaprojektowania specyficznych RNAi dla białek wirusa grypy: NP, PA, PB1. Wirus grypy typu A posiada wyjątkowo silny potencjał mutacyjny, dlatego cząsteczki siRNA, powinny być projektowane w konserwatywnych fragmentach genomu i stosowane w postaci mieszaniny różnorodnych siRNA [13,23].
Autor: Katarzyna Czuba
Literatura:
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje