

|
Zamknij X
|
 Elektroforezę kometkową charakteryzuje prostota wykonania, a także fakt, że z jej wykorzystaniem otrzymuje się powtarzalne wyniki badań. Z racji swego szerokiego zastosowania, comet assay należy do metod nieustannie modyfikowanych i ulepszanych. Tak więc, doczekała się już modyfikacji z wykorzystaniem przeciwciał albo hybrydyzacji z sondami molekularnymi (FISH), dzięki czemu możliwe jest szybkie i wybiórcze określenie zmian w obrębie DNA jądrowego [4].
Elektroforezę kometkową charakteryzuje prostota wykonania, a także fakt, że z jej wykorzystaniem otrzymuje się powtarzalne wyniki badań. Z racji swego szerokiego zastosowania, comet assay należy do metod nieustannie modyfikowanych i ulepszanych. Tak więc, doczekała się już modyfikacji z wykorzystaniem przeciwciał albo hybrydyzacji z sondami molekularnymi (FISH), dzięki czemu możliwe jest szybkie i wybiórcze określenie zmian w obrębie DNA jądrowego [4].
Metoda pozwala na analizę róznych typów uszkodzeń DNA, a także na badanie ich naprawy w indywidualnych komórkach. W 1978 roku Rydberg i Johanson, w trakcie prowadzonych badań wykazali, że jądra komórkowe poddawane lizie w warunkach alkalicznych pozostają stosunkowo skondensowanymi strukturami. Z kolei, po napromieniowaniu promieniowaniem jonizującym dochodzi dochodzi do rozluźnienia struktury DNA (co zależy od dawki promieniowania). Podstawą opisywanej metody stało się więc obserwowanie komórek po potraktowaniu ich 2M roztworem NaCl oraz detergentami anionowymi, które to prowadziły do powstawania „nukleoidów” zbudowanych z pętli DNA. Kilka lat później Ӧstling i Johanson (1984) wprowadzili etap krótkiego rozdziału elektroforetycznego komórek upzrednio poddanych lizie, a następnie zatopionych w agarozie na szkiełku mikroskopowym. Dalej, Singh i wsp. Zmodyfikowali całą procedurę metody i wprowadzili bufor alkaliczny do elektroforezy, a także dodatkowe rozwijanie DNA prowadzone bezpośrednio w alkalicznym roztworze elektroforetycznym. Zmiany te pozwalają na jednoczesną analizę pęknięć –jedno i –dwuniciowych, a także stanowią podstawę stosowanego testu kometkowego [8].
Choć metoda kometkowa (comet assay) jest stosunkowo prosta i szybka w wykonaniu, wymaga przeprowadzenia kilku kluczowych etapów, z których praktycznie każdy wymaga zastosowania innych buforów. W danych literaturowych pojawiają się rożne odmiany tego samego buforu (modyfikacje), które to powstają w trakcie wprowadzania zmian w pierwotnej metodologii. Jak już wspomniano, test kometkowy jest tak naprawdę modyfikowany od chwili wynalezienia jego pierwotnej wersji w 1978 roku (Rydberg i Johanson).
Bufory stosowane w metodzie kometkowej:
Ten wodny roztwór w składzie zawiera chlorek sodu oraz fosforan sodu, a dodatkowo może mieć także chlorek potasu oraz fosforan potasu. Głównym zadaniem PBS jest utrzymywanie stałe pH.Co więcej, stężenie jonów i ciśnienie osmotyczne buforu porównywalne jest do tego, jakie panuje w ludzkich płynach ustrojowych. Ze względu na dwie ważne cechy tj. izotoniczny charakter i brak toksyczność względem żywych komórek PBS powszechnie wykorzystywany jest w wielu róznych analizach [3].
Przygotowanie buforu PBS (w objętości 1 l):
W celu przygotowania roztworu PBS należy w 800 ml wody destylowanej kolejno rozpuścić:
Po zmieszaniu wszystkich składników ustala się pH roztworu do 7.4, używając roztworu kwasu solnego lub wodorotlenku sodu. Po ustaleniu pH bufor uzupełnić wodą destylowaną do objętości 1 litra. Przed użyciem należy poddać bufor autoklawowaniu (sterylizacji) [3].
W 50 ml probówce należy zmieszać: dichlorowodorek p-fenylenodiaminę (500 mg), 1x PBS (4,5 ml). Następnie, dodać około 400 µl 10 M NaOH, wkraplając do momentu aż pH roztworu osiągnie 7.5 – 8.0. Dodać 1x PBS, aby zwiększyć objętość do 5 ml i 45 ml glicerolu- do końcowej objętości równej 50 ml. Całość przygotowanej próbki dokładnie zvorteksować,a w razie konieczności nanosić w objętości 10 µl na próbkę. Przygotowany bufor można przechowywać do jednego miesiąca w temperaturze -20°C, a w trakcie przechowywania może wystąpić ciemnienie roztworu [7].
Przygotowanie fragmentów tkanek do analizy
Mały kawałek tkanki, która ma zostać poddana analizie w teście kometkowym, należy umieścić w 1-2 ml lodowatego 1x stężonego buforu PBS (wolnego od jonów Mg2+ i Ca2+) z dodatkiem 20 mM EDTA. Za pomocą skalpela pociąć tkankę na bardzo małe fragmenty, po czym pozostawić na 5 minut. Na tym etapie liczy się komórki, wiruje i ponownie rozpuszcza otrzymaną zawiesinę do 105 komórek/ml w lodowatym (wolnym od jonów magnezu i wapnia) PBS (1x). Następnie, do próbki dodać 1-2 ml lodowatego 20 mM roztworu EDTA w 1x PBS, tkankę zmielić na bardzo małe kawałki i pozostawić do odstania na 5 minut. Odzyskanie zawiesiny komórek pozwoli na uniknięcie przenoszenia zanieczyszczeń. Na tym etapie ponownie zlicza się komórki i zawiesza ich osad w 1xPBS (jak wyżej) [7].
Przygotowanie preparatów do comet assay [1],[2].
Olive P.L. i Banáth J.P.(2006), przedstawili wykorzystanie metody kometkowej do pomiaru uszkodzenia DNA w poszczególnych komórkach eukariotycznych. Choć większość badań opiera się na wykorzystaniu metody kometkowej do pomiaru przerw w pojedynczej nici DNA, wprowadzenie odpowiednich modyfikacji w metodzie umożliwia wykrywanie przerw również w podwójnej nici DNA, połączeń krzyżowych czy jąder apoptotycznych. Czułość testu w przeprowadzonym badaniu wynosiła około 50 pęknięć nici DNA na diploidalną komórkę ssaków. Ponadto, metoda ta znalazła także zastosowanie do komórek drożdży, pierwotniaków czy roślin. Przedstawiony przez Olive P.L. i Banáth J.P.(2006) protokół może być zakończony w mniej niż 24 godziny, co potwierdza,że comet assay jest stosunkowo szybką metodą analizy [5].
Analiza komórek apoptotycznych
W trakcie procedu apoptozy dochodzi do uszkodzenia (degradacji) komórkowego DNA. Degradacja zachodzi pod wpływem enzymu endonukleazy, która to aktywowana jest przez kaspazy. W trakcie tego procesu DNA hydrolizowane jest preferencyjnie w miejscach pomiędzy nukleosomami. Na skutek degradacji DNA rozpada się na fragmenty o długości ok 180 par zasad bądź ich wielokrotności (tj. 360pz, 560pz itd). W pzreprowadzonych badaniach wykazano, że ilość powstających fragmentów DNA zależna jest od tego, w jakich zachodzi komórkach, tj. czy są to komórki apoptotyczne czy nekrotyczne. Fragmentacja dla komórek apoptotycznych jest wyższa niż dla komórek nekrotycznych, na czym też test kometkowy się opiera. Comet assay umożliwia odróżnienie dwóch typów śmierci komórek (apoptotycznej i nekrotycznej) właśnie na tej podstawie [6].
Dla komórek nekrotycznych i apoptotycznych wykonuje się w identyczny sposób jak dla wszystkich innych typów komórek. Naniesione i utrwalone na szkiełku komórki poddaje się rozdziałowi elektroforetycznemu, podczas którego (po przyłożeniu napięcia pofragmentowane DNA opuszcza komórkę i migruje w żelu w kierunku anody). Po zakończeniu rozdziału elektroforetycznego, DNA wybarwia się w celu jego uwidocznienia . Najczęściej stosuje się w tym celu bromek etydyny. DNA, które opuściło komórkę po uwidocznieniu tworzy charakterystyczny wzór- podobny do ogona komety, zaś to pozostałe w komórce wygląda jak głowa komety. W przypadku, gdy nie nie doszło do pęknięć lub cięcia DNA, obserwuje się świecenie tylko w obrębie komórki tj. głowy komety. Wszystko wynika z faktu, że nie pocięte DNA jest zbyt duże, by pod wpływem pola elektrycznego opuścić komórkę, w związku z czym pozostaje uwięzione w strukturach komórkowych. Komórki nekrotyczne również dają fluoryzujący ogon, aczkolwiek precyzyjny pomiar parametrów, które dokładnie opisują intensywność świecenia ogona pozwala na ich odróżnienie od komórek apoptotycznych [6].
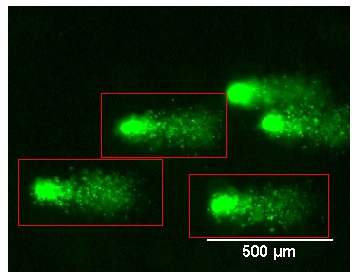
Zdjęcie: Kometki, http://library.wolfram.com/examples/cometassay/
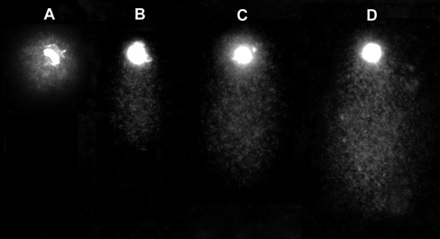
Zdjęcie: Komórki HeLa po analizie comet assay, http://jhc.sagepub.com/content/59/7/655/F2.expansion.html
Wojewódzka M. i wsp. (2000) przeprowadzili test kometkowy na komórkach krwi (limfocytach) pobranych z żyły. W badaniach wykorzystano znacznie skróconą metologię (w porónaniu do opisywanych w literaturze procedurami), dzięki czemu uproszczono i znacznie skrócono poszczególne etapy testu. W trakcie doświadczenia uzyskano dobrą korelację pomiędzy wielkością uszkodzenia (mierzoną mierzoną tzw. „momentem ogonowym”), a zastosowaną do badań dawką promieniowania jonizującego. Pomimo wprowadzonych przez badaczy modyfikacji w metodzie dowiedzono, że kinetyk anaprawy podwójnoniciowych pęknięć DNA jest jak najbardziej zgodna z podawanymi danymi literaturowymi [8].
Liza komórek krwi (limfocytów) do testu kometkowego (procedura wg Wojewódzka M. i wsp., 2000):
Przed rozpoczęciem tego etapu przygotowano podstawowy bufor do lizy (tj.: 2,5 M NaCl, 100mM EDTA, 10 mM Tris-HCl, 1% sarkozyl-N-laurylosarkozyna, bufor o pH=9.5). Bezpośrednio przed użyciem do buforu dodano 0,5% Triton X-100 oraz 10% DMSO (dimetylosulfotlenek). Całość mieszano z wykorzystaniem mieszadłą magnetycznego (około 20 minut) i schładzano w lodówce. Do tak przygotowanego zimnego buforu wkładano szkiełka z naniesionymi na nie komórkami zawieszonymi w agarozie. Etap lizy prowadzono w ciemności przez 1-2 godzin w temperaturze 4°C.
Płukanie preparatów po lizie (procedura wg Wojewódzka M. i wsp., 2000):
Preparaty przemywano 3-krotnie z wykorzystaniem buforu do elektroforezy (tj.: 300 mM octan sodowy, 100 mM Tris-HCl o pH=8.5). Preparaty pozostawiono w świeżej porcji buforu na co najmniej 1 godzinę, w celu dokładnego wypłukania resztek soli (pozostałych po etapie lizy).
Rozdział elektroforetyczny (procedura wg Wojewódzka M. i wsp., 2000):
W aparacie do elektroforezy umieszczono wypłukane szkiełka podstawowe, a rozdział prowadzono przy napięciu 14V i natężeniu 11-12 mM, przez 1 godzinę w temperaturze nie przekraczającej 10°C. Cały rozdział prowadzono w ciemności. Po skończonym rozdziale szkiełka zostały 3 razy przepłukane w w buforze neutralizującym (tj.: 0,4 M Tris o pH= 7.5). Szkiełka następnie osuszono z nadmiaru roztworu i poddano barwieniu (1µM roztworem DAPI), po czym nakryto preparat szkiełkiem nakrywkowym, po czym preparaty włożono do pudełka wyłożonego wilgotną bibułą i przechowywano w lodówce przez około 20 godzin. Przygotowane w ten sposób preparaty mikroskopowe poddawano następnie analizie komputerowej[8].
Wprowadzone przez Wojewódzką M. i wsp. (2000) modyfikacje w metodzie kometkowej, polegające na prowadzeniu testu w pH obojętnym, pozwoliły na otrzymanie komet o bardziej zwartym zarysie, niż w testach wykonywanych w publikacjach innych autorów. Wcześniej opisane procedury (przed modyfikacją powyższych autorów) dostarczały „obrazy” komet o bardzo wydłużónych i rozmytych ogonach, co obniżało precyzyjnośc pomiarów, a momentami wręcz uniemożliwiało ich przeprowadzenie [8].
Analiza komet metodą Gedika
Do oceny skali uszkodzenia materiału genetycznego po teście kometkowym zazwyczaj wykorzystuje się specjalistyczne oprogramowanie do komputerowej analizy obrazu. Innym, prostym sposobem pozwalającym na analize obrazu komet jest tzw. metoda Gedika i wsp. (1992), która opiera się na klasyfikacji uszkodzeń jądrowego DNA w komórce do pięciu klas.
Skala ta obejmuje charakterystyczne oznaczenia:
1) N –brak uszkodzeń (mniej niż 5% DNA w ogonie komety)
2) L-małe uszkodzenia (5- 25% DNA w ogonie komety)
3) M- średnie uszkodzenia (20-40% DNA w ogonie komety)
4) H- duże uszkodzenia (40 – 95% DNA w ogonie komety)
5) T-całkowite uszkodzenia (ponad 95% DNA w ogonie komety)[4].
Autor: Lidia Koperwas
Literatura:
[1]. http://www.kucharczyk.com.pl/instrukcje/poradykometa.pdf . Dr Marcin Schmidt, Uniwersytet Przyrodniczy w Poznaniu, http://www.up.poznan.pl/~mschmidt/
[2]. http://www.kucharczyk.com.pl/
[3]. http://www.e-biotechnologia.pl/Artykuly/sol-fizjologiczna
[4].Brzuzan P., Woźny M., Łuczyński M.K., 2007. Toksykologia molekularna, przewodnik do ćwiczeń, Olsztyn 2001. http://biotechnology.keyland.biz/pdf/tox_mol_updated.pdf
[5]. Olive P.L. i Banáth J.P., 2006. The comet assay: a method to measure DNA damage in individual cells. http://www.ncbi.nlm.nih.gov/pubmed/17406208 (abstract)
[6]. http://bioinfo.mol.uj.edu.pl/articles/Kacprzyk04
[7]. http://www.amsbio.com/protocols/Comet_Assay.pdf
[8]. Wojewódzka M., Grądzka I, Buraczewska I., 2000. Test kometowy w pH obojętnym. Instytut Chemii i Techniki Jądrowe, Warszawa 32/28. Raporty IchTJ.Seria B nr 2/2000. http://www.iaea.org/inis/collection/NCLCollectionStore/_Public/32/032/32032373.pdf
25 maja 2018 roku zacznie obowiązywać Rozporządzenie Parlamentu Europejskiego i Rady (UE) 2016/679 z dnia 27 kwietnia 2016 r (RODO). Potrzebujemy Twojej zgody na przetwarzanie Twoich danych osobowych przechowywanych w plikach cookies. Poniżej znajdziesz pełny zakres informacji na ten temat.
Zgadzam się na przechowywanie na urządzeniu, z którego korzystam tzw. plików cookies oraz na przetwarzanie moich danych osobowych pozostawianych w czasie korzystania przeze mnie ze strony internetowej Laboratoria.net w celach marketingowych, w tym na profilowanie i w celach analitycznych.
Administratorami Twoich danych będziemy my: Portal Laboratoria.net z siedzibą w Krakowie (Grupa INTS ul. Czerwone Maki 55/25 30-392 Kraków).
Chodzi o dane osobowe, które są zbierane w ramach korzystania przez Ciebie z naszych usług w tym zapisywanych w plikach cookies.
Przetwarzamy te dane w celach opisanych w polityce prywatności, między innymi aby:
dopasować treści stron i ich tematykę, w tym tematykę ukazujących się tam materiałów do Twoich zainteresowań,
dokonywać pomiarów, które pozwalają nam udoskonalać nasze usługi i sprawić, że będą maksymalnie odpowiadać Twoim potrzebom,
pokazywać Ci reklamy dopasowane do Twoich potrzeb i zainteresowań.
Zgodnie z obowiązującym prawem Twoje dane możemy przekazywać podmiotom przetwarzającym je na nasze zlecenie, np. agencjom marketingowym, podwykonawcom naszych usług oraz podmiotom uprawnionym do uzyskania danych na podstawie obowiązującego prawa np. sądom lub organom ścigania – oczywiście tylko gdy wystąpią z żądaniem w oparciu o stosowną podstawę prawną.
Masz między innymi prawo do żądania dostępu do danych, sprostowania, usunięcia lub ograniczenia ich przetwarzania. Możesz także wycofać zgodę na przetwarzanie danych osobowych, zgłosić sprzeciw oraz skorzystać z innych praw.
Każde przetwarzanie Twoich danych musi być oparte na właściwej, zgodnej z obowiązującymi przepisami, podstawie prawnej. Podstawą prawną przetwarzania Twoich danych w celu świadczenia usług, w tym dopasowywania ich do Twoich zainteresowań, analizowania ich i udoskonalania oraz zapewniania ich bezpieczeństwa jest niezbędność do wykonania umów o ich świadczenie (tymi umowami są zazwyczaj regulaminy lub podobne dokumenty dostępne w usługach, z których korzystasz). Taką podstawą prawną dla pomiarów statystycznych i marketingu własnego administratorów jest tzw. uzasadniony interes administratora. Przetwarzanie Twoich danych w celach marketingowych podmiotów trzecich będzie odbywać się na podstawie Twojej dobrowolnej zgody.
Dlatego też proszę zaznacz przycisk "zgadzam się" jeżeli zgadzasz się na przetwarzanie Twoich danych osobowych zbieranych w ramach korzystania przez ze mnie z portalu *Laboratoria.net, udostępnianych zarówno w wersji "desktop", jak i "mobile", w tym także zbieranych w tzw. plikach cookies. Wyrażenie zgody jest dobrowolne i możesz ją w dowolnym momencie wycofać.
Więcej w naszej POLITYCE PRYWATNOŚCI
Recenzje